Título de la obra:
Bacterias: Escherichia coli
Autores:
Juan Esteban Rivera García
Juan Guillermo Rivera Berrío
Código JavaScript para el libro: Joel Espinosa Longi, IMATE, UNAM.
Recursos interactivos: DescartesJS
Fuentes: Lato y UbuntuMono
Imagen de portada: E coli a 10000x (HandWiki, CC BY-SA 3.0)
Red Educativa Digital Descartes
Córdoba (España)
descartes@proyectodescartes.org
https://proyectodescartes.org
Proyecto iCartesiLibri
https://proyectodescartes.org/iCartesiLibri/index.htm
ISBN: 978-84-18834-84-4

Esta obra está bajo una licencia Creative Commons 4.0 internacional: Reconocimiento-No Comercial-Compartir Igual.
La bacteria Escherichia coli (E. coli) es un microorganismo que ha sido ampliamente estudiado e investigado debido a su papel en diversas enfermedades intestinales y extraintestinales en humanos y animales
E. coli es una bacteria gram negativa, anaerobia predominante en la flora intestinal humana, y normalmente se mantiene inocua y confinada al lumen intestinal. Sin embargo, en huéspedes inmunosuprimidos o cuando las barreras gastrointestinales son sobrepasadas, las cepas de E. coli pueden producir infecciones.
Dentro de las infecciones más frecuentes originadas por E. coli se encuentran las enfermedades entéricas y diarreas. Las cepas de E. coli que causan diarreas producen diversos factores de virulencia, como el factor de virulencia verotoxígeno (stx) y el factor de virulencia enterotoxígeno (elt).
En este libro abordaremos los siguientes temas:
Este libro podría ser de interés para los estudiantes de la salud, los profesionales de la salud, los investigadores y los interesados en la enfermedad intestinal.
Las bacterias están con nosotros en todas partes y en todo momento interactuamos con ellas, hazte unas preguntas antes de sumergirte de lleno a este fascinante y amplio mundo de las bacterias.
¿Qué son las bacterias?
¿Cuántas bacterias conoces?
¿Por qué enferman las bacterias?
¿Qué sabes de la bacteria E. coli?
Todas esas preguntas y muchas más serán respondidas en los capítulos de este libro.
Para ilustrar mejor la información suministrada en este libro, hemos incluido, entre otros elementos multimedia, los siguientes:

Bacterias, imagen generada por la IA DALL-E 3.
Las bacterias son organismos procariotas unicelulares, que se encuentran en casi todas las partes de la Tierra. Son vitales para los ecosistemas del planeta. Algunas especies pueden vivir en condiciones realmente extremas de temperatura y presión. El cuerpo humano está lleno de bacterias, de hecho se estima que contiene más bacterias que células humanas. La mayoría de bacterias que se encuentran en el organismo no producen ningún daño, al contrario, algunas son beneficiosas. Una cantidad relativamente pequeña de especies son las que causan enfermedades
Las bacterias son microorganismos que pueden tener distintas formas. Pueden ser esféricas, alargadas o espirales. Existen bacterias perjudiciales, llamadas patogénicas, las cuales causan enfermedades; pero también hay bacterias buenas. Por ejemplo, en nuestro sistema digestivo, en el intestino, tenemos bacterias que son muy necesarias para que nuestro cuerpo funcione correctamente. Lo más sorprendente sobre las bacterias es que en nuestro cuerpo tenemos 10 veces más células bacterianas que células humanas. Las bacterias también son muy importantes para la biotecnología (Ibid.).
La palabra bacteria es el plural de la bacteria neolatina, que es la latinización del griego antiguo βακτήριον ( baktḗrion ), el diminutivo de βακτηρία ( baktēría ), que significa "bastón", porque las primeras que se descubrieron tenían forma de varilla
Los antepasados de las bacterias fueron microorganismos unicelulares que fueron las primeras formas de vida que aparecieron en la Tierra, hace unos 4 mil millones de años. Durante aproximadamente 3 mil millones de años, la mayoría de los organismos eran microscópicos, y las bacterias y arqueas eran las formas de vida dominantes
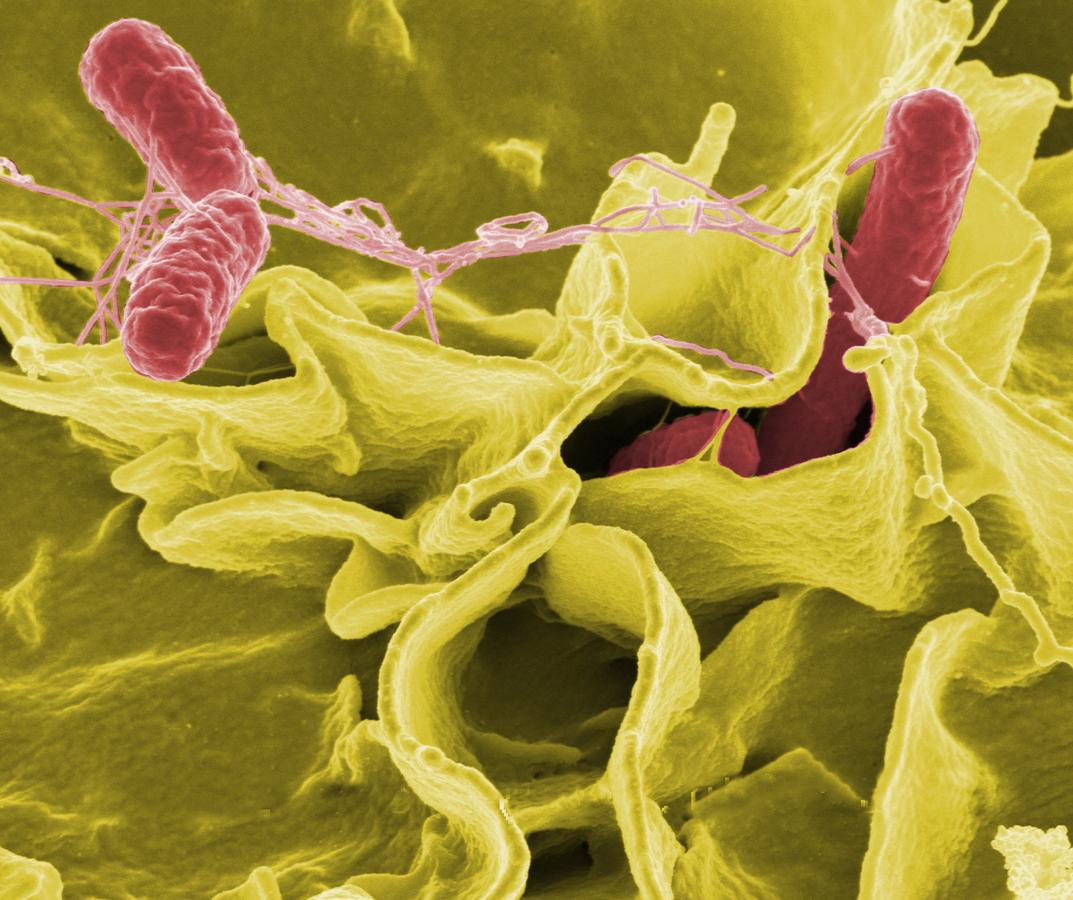
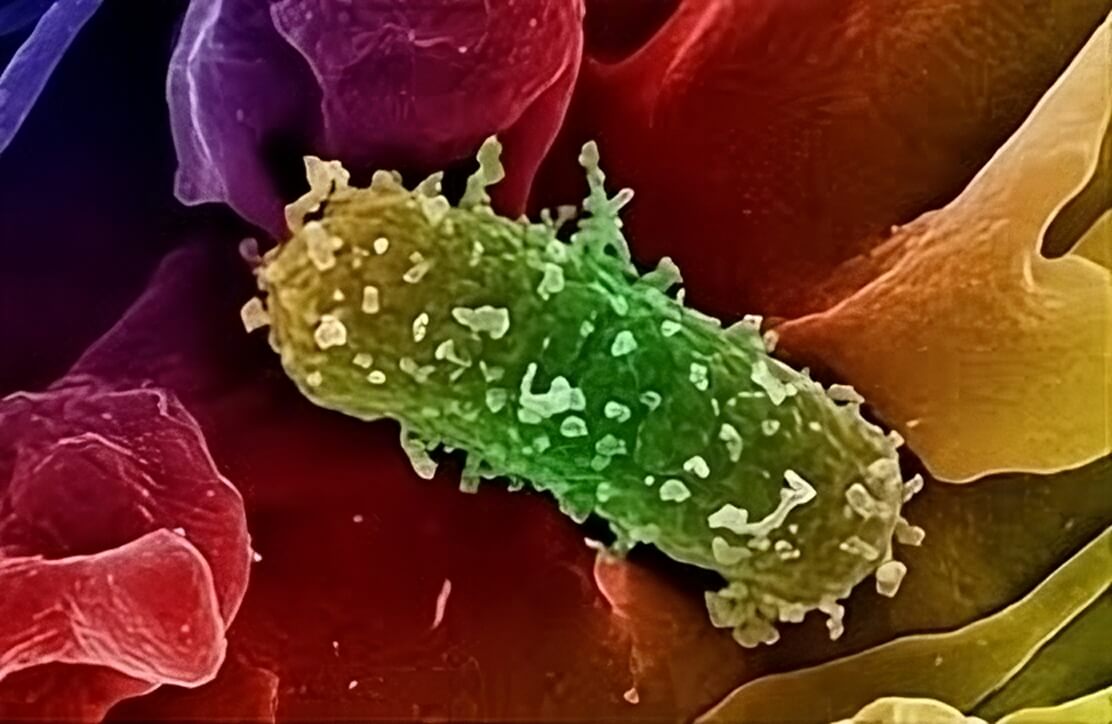
Micrografía electrónica de barrido coloreada que muestra Klebsiella pneumoniae resistente a carbapenémicos interactuando con un neutrófilo, imagen de HandWiki, CC BY-SA 3.0.
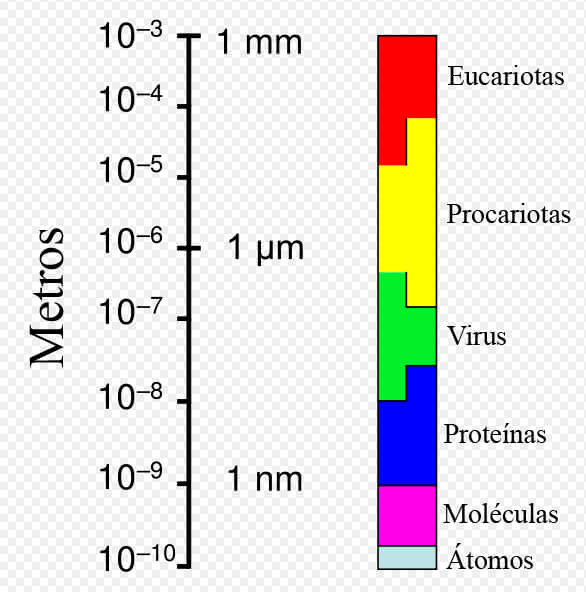
Tamaño. Las bacterias muestran una amplia diversidad de formas y tamaños. Las células bacterianas tienen aproximadamente una décima parte del tamaño de las células eucariotas y suelen tener entre 0,5 y 5,0 micrómetros de longitud. Sin embargo, algunas especies son visibles a simple vista; por ejemplo, Thiomargarita namibiensis mide hasta medio milímetro de largo, Epulopiscium fishelsoni alcanza 0,7 mm, y Thiomargarita magnifica puede alcanzar incluso 2 cm de largo, lo que es 50 veces más grande que otras bacterias conocidas
Forma. La mayoría de las especies bacterianas son esféricas, llamadas cocos (singular cocos, del griego kókkos, grano, semilla), o tienen forma de bastón, llamadas bacilos (sing . bacillus, del latín baculus, palo). Algunas bacterias, llamadas vibrio, tienen forma de bastones ligeramente curvados o de coma; otras pueden tener forma de espiral, llamados espirilos o en forma de sacacorchos, llamadas espiroquetas. Se han descrito un pequeño número de otras formas inusuales, como bacterias con forma de estrella
Multicelularidad. La mayoría de las especies bacterianas existen como células individuales; otros se asocian en patrones caracte- rísticos: Neisseria en forma de diplococos (pares), los estrep- tococos forman cadenas y los estafilococos se agrupan en "racimos de uvas". Las bacterias también pueden agruparse para formar estructuras multicelulares más grandes, como los filamentos alargados de las especies Actinomycetota, los agregados de las especies Myxobacteria y Streptomyces. Estas estructuras multicelulares a menudo solo se ven en ciertas condiciones. Por ejemplo, cuando carecen de aminoácidos, las mixobacterias detectan las células circundantes en un proceso conocido como detección de quórum, migran unas hacia otras y se agregan para formar cuerpos fructíferos de hasta 500 micrómetros de largo y que contienen aproximadamente 100.000 células bacterianas
Biopelículas. Las bacterias a menudo se adhieren a las superficies y forman agregaciones densas llamadas biopelículas, y formaciones más grandes conocidas como tapetes microbianos
de espesor hasta medio metro de profundidad, y pueden contener múltiples especies de bacterias, protistas y arqueas. Las bacterias que viven en biopelículas muestran una disposición compleja de células y componentes extracelulares, formando estructuras secundarias, como microcolonias, a través de las cuales existen redes de canales para permitir una mejor difusión de nutrientes
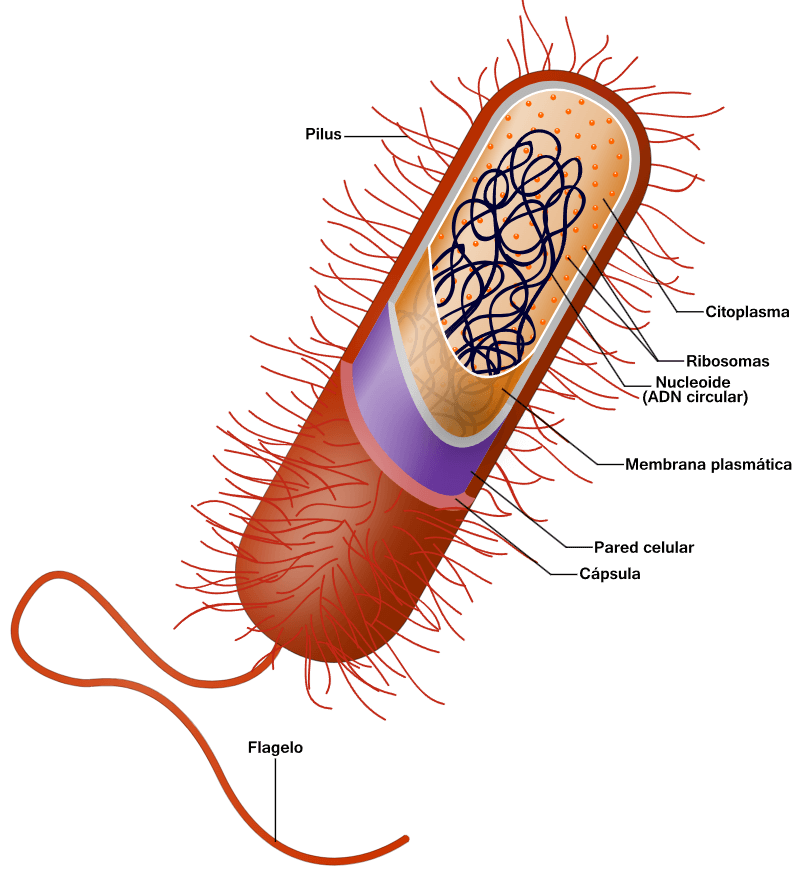
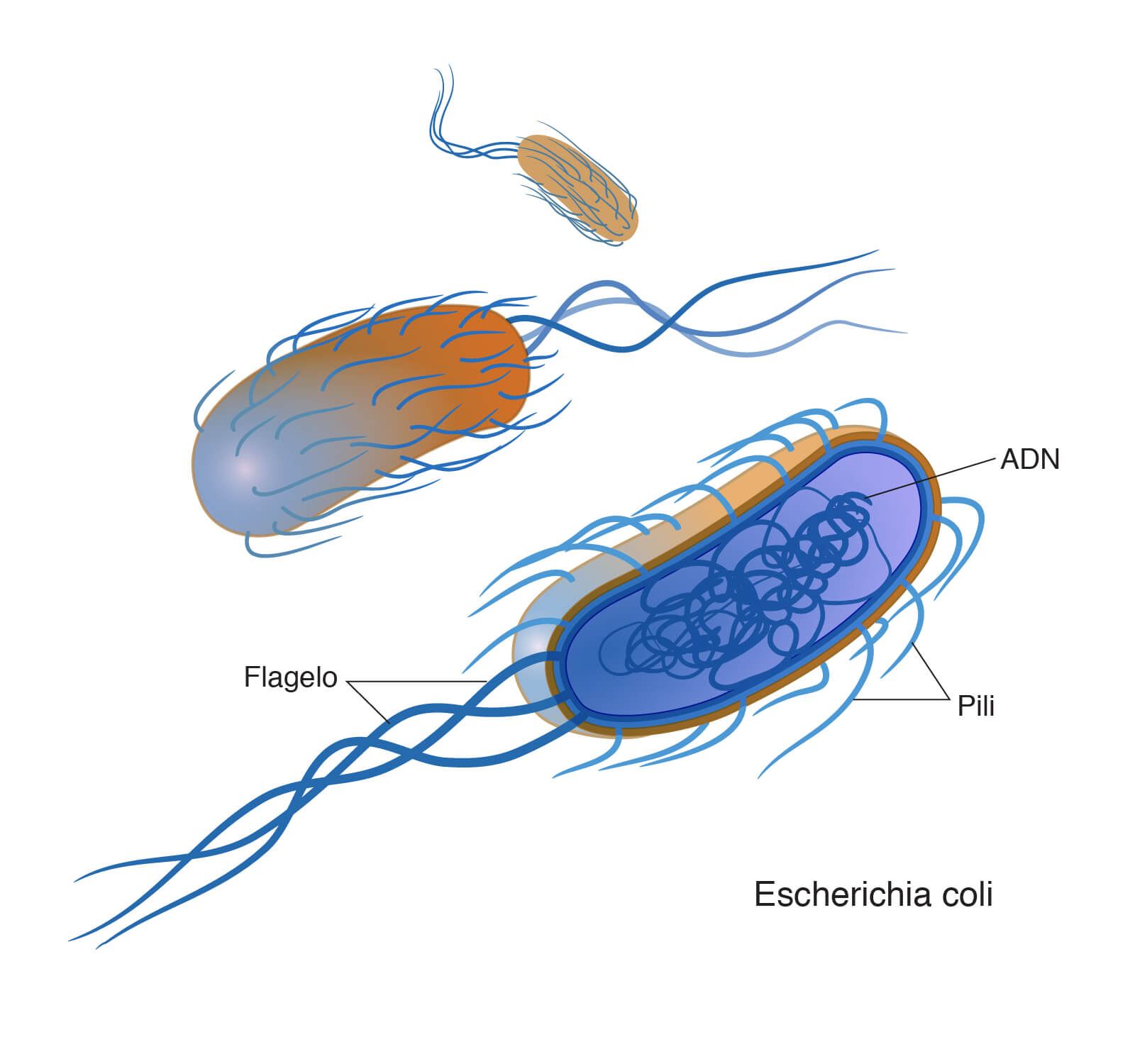
La célula bacteriana está rodeada por una membrana celular compuesta principalmente de fosfolípidos. Esta membrana encierra el contenido de la célula y actúa como una barrera para mantener nutrientes, proteínas y otros componentes esenciales del citoplasma dentro de la célula. A diferencia de las células euca- riotas, las bacterias generalmente carecen de grandes estructuras unidas a membranas en su citoplasma, como el núcleo, las mitocondrias, los cloroplastos y otros orgánulos presentes en las células eucariotas
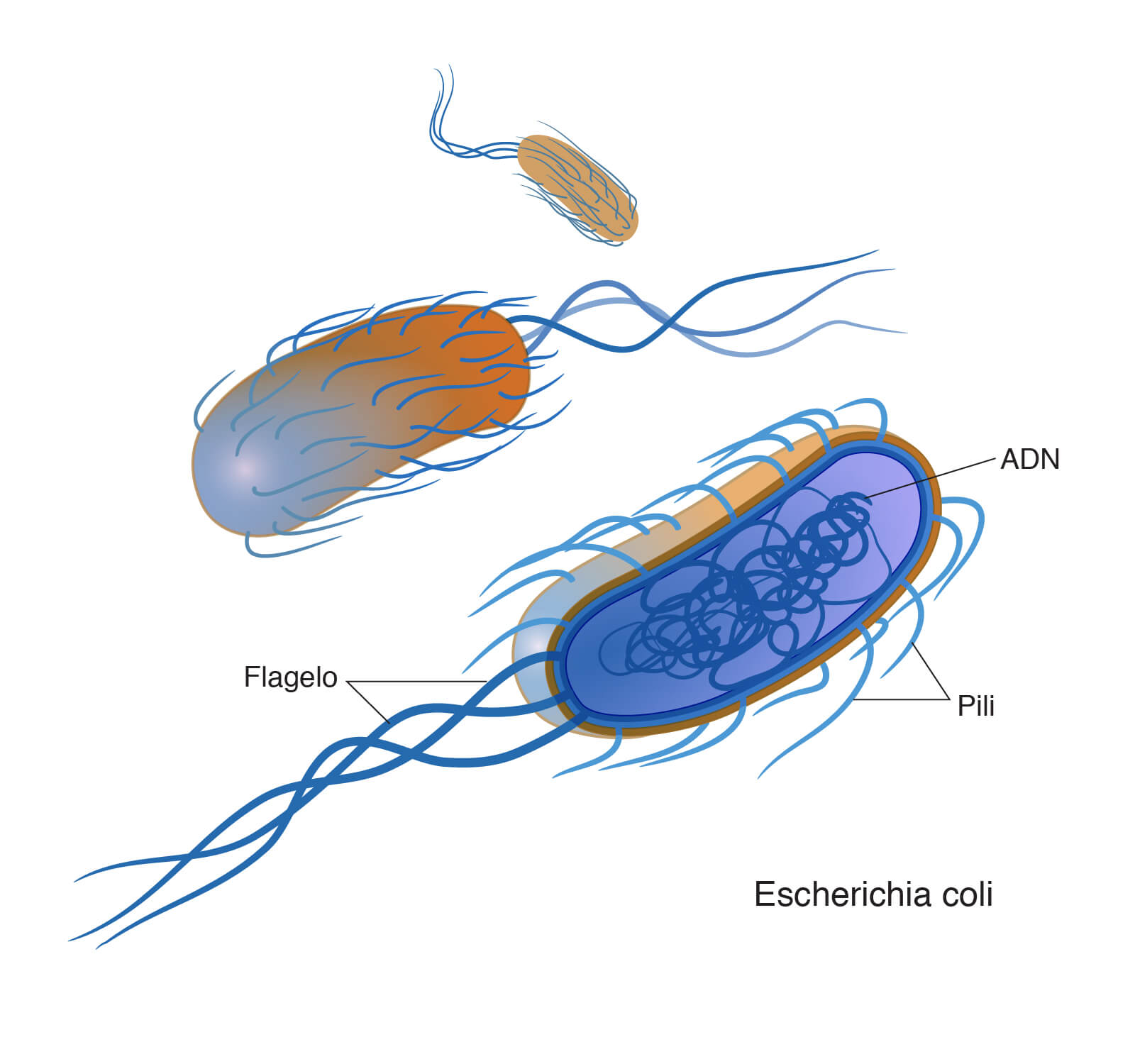
Las bacterias no tienen un núcleo rodeado de membranas y su material genético suele ser un único cromosoma bacteriano circular de ADN ubicado en el citoplasma en un cuerpo de forma irregular llamado nucleoide
bacterias contienen ribosomas para la producción de proteínas, pero la estructura del ribosoma bacteriano es diferente a la de las eucariotas y las arqueas. Algunas bacterias producen gránulos de almacenamiento de nutrientes intracelulares, como glucógeno
Alrededor del exterior de la membrana celular se encuentra la pared celular. Las paredes celulares bacterianas están hechas de peptidoglicano (también llamado mureína), que está hecho de cadenas de polisacáridos entrecruzadas por péptidos que contienen D- aminoácidos. Las paredes celulares bacterianas son diferentes de las paredes celulares de plantas y hongos, que están hechas de celulosa y quitina, respectivamente
En términos generales, existen dos tipos diferentes de pared celular en las bacterias, que las clasifican en bacterias Gram positivas y bacterias Gram negativas. Los nombres provienen de la reacción de las células a la tinción de Gram, una prueba de larga data para la clasificación de especies bacterianas.
Las bacterias Gram positivas poseen una pared celular gruesa que contiene muchas capas de peptidoglicano y ácidos teicoicos. Por el contrario, las bacterias Gram negativas tienen una pared celular relativamente delgada que consta de unas pocas capas de peptidoglicano rodeadas por una segunda membrana lipídica que contiene lipopolisacáridos y lipoproteínas. La mayoría de las bacterias tienen la pared celular Gram-negativa, y sólo los miembros del grupo Bacillota y actinomycetota (anteriormente conocidos como bacterias Gram-positivas de bajo G+C y alto G+C, respectivamente) tienen la disposición alternativa Gram-positiva
producir diferencias en la susceptibilidad a los antibióticos; por ejemplo, la vancomicina puede matar sólo bacterias Gram positivas y es ineficaz contra patógenos Gram negativos, como Haemophilus influenzae o Pseudomonas aeruginosa. Algunas bacterias tienen estructuras de pared celular que no son clásicamente Gram-positivas ni Gram-negativas. Esto incluye bacterias clínicamente importantes, como las micobacterias, que tienen una pared celular gruesa de peptidoglicano como una bacteria Gram-positiva, pero también una segunda capa externa de lípidos
En muchas bacterias, una capa S de moléculas de proteínas dispuestas rígidamente cubre el exterior de la célula. Esta capa proporciona protección química y física para la superficie celular y puede actuar como una barrera de difusión macromolecular. Las capas S tienen diversas funciones y se sabe que actúan como factores de virulencia en especies de Campylobacter y contienen enzimas de superficie en Bacillus stearothermophilus
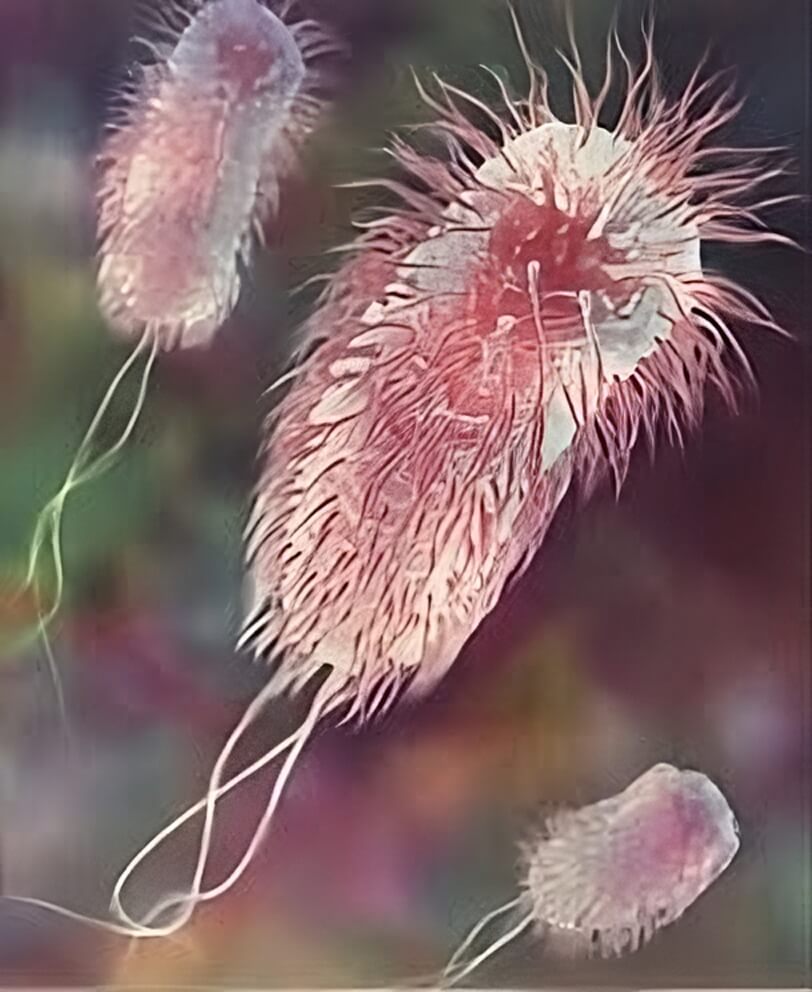
Los flagelos son estructuras proteicas rígidas, de unos 20 nanómetros de diámetro y hasta 20 micrómetros de longitud, que se utilizan para la motilidad . Los flagelos son impulsados por la energía liberada por la transferencia de iones a lo largo de un gradiente electroquímico a través de la membrana celular.
Las fimbrias (a veces llamadas "pili de unión") son finos filamentos de proteína, generalmente de 2 a 10 nanómetros de diámetro y hasta varios micrómetros de longitud. Se distribuyen por la superficie de la célula y se asemejan a pelos finos cuando se observan al microscopio electrónico. Se cree que las fimbrias participan en la unión a superficies sólidas o a otras células, y son esenciales para la virulencia de algunos patógenos bacterianos. Los pili (sing . pilus) son apéndices celulares, un poco más grandes que las fimbrias, que pueden transferir material genético entre células bacterianas en un proceso
llamado conjugación , donde se denominan pili de conjugación o pili sexual (ver genética bacteriana, más abajo). También pueden generar movimiento donde se les llama pili tipo IV
Muchas bacterias producen el glicocáliz para rodear sus células y su complejidad estructural varía: desde una capa viscosa desorganizada de sustancias poliméricas extracelulares hasta una cápsula altamente estructurada. Estas estructuras pueden proteger a las células de la absorción por células eucariotas como los macrófagos (parte del sistema inmunológico humano). También pueden actuar como antígenos y participar en el reconocimiento celular, además de ayudar a la adhesión a superficies y la formación de biopelículas.
El ensamblaje de estas estructuras extracelulares depende de los sistemas de secreción bacteriana. Estos transfieren proteínas desde
el citoplasma al periplasma o al entorno que rodea la célula. Se conocen muchos tipos de sistemas de secreción y estas estructuras suelen ser esenciales para la virulencia de los patógenos, por lo que se estudian intensamente
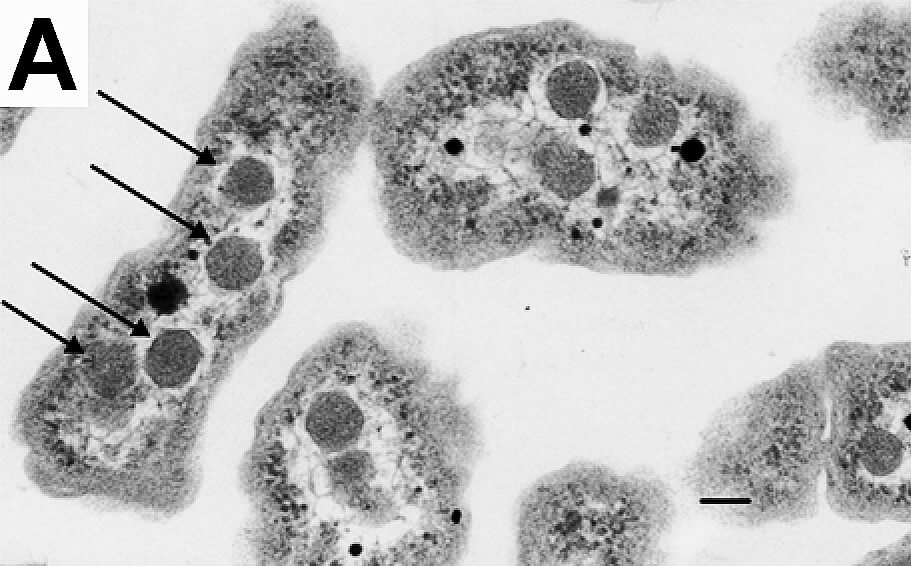
Algunos géneros de bacterias Gram positivas, como Bacillus, Clostridium, Sporohalobacter, Anaerobacter y Heliobacterium, pueden formar estructuras latentes altamente resistentes llamadas endosporas
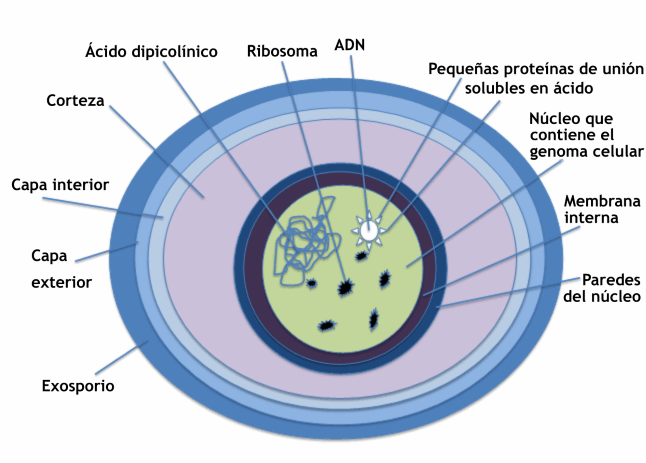
Las endosporas no muestran un metabolismo detectable y pueden sobrevivir a tensiones físicas y químicas extremas, como altos niveles de luz ultravioleta, radiación gamma, detergentes, desinfectantes, calor, congelación, presión y desecación
Las bacterias formadoras de endosporas pueden causar enfermedades; por ejemplo, el ántrax puede contraerse mediante la inhalación de endosporas de Bacillus anthracis, y la contaminación de heridas punzantes profundas con endosporas de Clostridium tetani provoca tétanos, que, al igual que el botulismo, es causado por una toxina liberada por las bacterias que crecen a partir de las esporas. La infección por Clostridioides difficile, un problema común en entornos de atención médica, es causada por bacterias formadoras de esporas

"Cultivo de muchas bacterias de diferente forma", Imagen creada por la IA Lexica.art

A diferencia de los organismos multicelulares, el aumento del tamaño celular (crecimiento celular) y la reproducción por división celular están estrecha- mente relacionados en los organismos unicelulares. Las bacterias crecen hasta un tamaño fijo y luego se reproducen mediante fisión binaria, una forma de reproducción asexual
En el laboratorio, las bacterias suelen cultivarse en medios sólidos o líquidos. Los medios de crecimiento sólidos, como las placas de agar, se utilizan para aislar cultivos puros de una cepa bacteriana. Sin embargo, los medios de crecimiento líquidos se utilizan cuando se
requiere la medición del crecimiento o grandes volúmenes de células. El crecimiento en medios líquidos agitados se produce como una suspensión celular uniforme, lo que hace que los cultivos sean fáciles de dividir y transferir, aunque es difícil aislar bacterias individuales de medios de enriquecimiento. El uso de medios selectivos (medios con nutrientes específicos agregados o deficientes, o con antibióticos agregados) puede ayudar a identificar organismos específicos
La mayoría de las técnicas de laboratorio para el cultivo de bacterias utilizan altos niveles de nutrientes para producir grandes cantidades de células de forma económica y rápida. Sin embargo, en ambientes naturales, los nutrientes son limitados, lo que significa que las bacterias no pueden continuar reproducién- dose indefinidamente. Esta limitación de nutrientes ha llevado a la evolución de diferentes estrategias de crecimiento. Algunos organismos pueden crecer extremadamente rápido cuando hay nutrientes disponibles, como la formación de floraciones de algas y cianobacterias que a menudo ocurren en los lagos durante el verano
El crecimiento bacteriano sigue cuatro fases. Cuando una población de bacterias ingresa por primera vez a un entorno rico en nutrientes que permite el crecimiento, las células necesitan adaptarse a su nuevo entorno. La primera fase de crecimiento es la fase de retraso o adaptación, un período de crecimiento lento cuando las células se adaptan al entorno rico en nutrientes y se preparan para un crecimiento rápido. La fase de retraso tiene altas tasas de biosíntesis, ya que se producen las proteínas necesarias para un crecimiento rápido
La fase logarítmica está marcada por un rápido crecimiento exponencial. La velocidad a la que las células crecen durante esta fase se conoce como tasa de crecimiento (k), y el tiempo que tardan las células en duplicarse se conoce como tiempo de generación (g). Durante la fase logarítmica, los nutrientes se metabolizan a máxima velocidad hasta que uno de los nutrientes se agota y comienza a limitar el crecimiento.
La tercera fase de crecimiento es la fase estacionaria y es causada por el agotamiento de los nutrientes. Las células reducen su actividad metabólica y consumen proteínas celulares no esenciales. La fase estacionaria es una transición de un crecimiento rápido a un estado de respuesta al estrés y hay una mayor expresión de genes implicados en la reparación del ADN, el metabolismo antioxidante y el transporte de nutrientes. La fase final es la fase de muerte donde las bacterias se quedan sin nutrientes y mueren.
En la siguiente página, presentamos la imagen del Staphylococcus aureus (Imagen descargada de Wikipedia). Pero, para ver la imagen deber armar el puzle, el cual tiene 25 piezas. Puedes ampliarlo a pantalla completa.
La mayoría de las bacterias tienen un único cromosoma circular que puede variar en tamaño desde sólo 160.000 pares de bases en la bacteria endosimbiótica Carso- nella ruddii, hasta 12.200.000 pares de bases (12,2 Mbp) en la bacteria que habita en el suelo Sorangium cellulosum
Las bacterias, como organismos asexuales, heredan una copia idéntica del genoma de sus padres y son clonales. Sin embargo, todas las bacterias pueden evolucionar mediante selección sobre cambios en su material genético (ADN) causados por recombinación genética o mutaciones. Las mutaciones surgen de errores cometidos durante la replicación del ADN o de la exposición a mutágenos. Las tasas de
mutación varían ampliamente entre diferentes especies de bacterias e incluso entre diferentes clones de una sola especie de bacteria. Los cambios genéticos en los genomas bacterianos surgen de una mutación aleatoria durante la replicación o de una "mutación dirigida por estrés", donde los genes involucrados en un proceso particular de limitación del crecimiento tienen una mayor tasa de mutación
Algunas bacterias transfieren material genético entre células. Esto puede ocurrir de tres maneras principales. Primero, las bacterias pueden absorber ADN exógeno de su entorno en un proceso llamado transformación. Muchas bacterias pueden absorber ADN del medio ambiente de forma natural, mientras que otras deben ser alteradas químicamente para inducirlas a absorber ADN
En circunstancias normales, la transducción, conjugación y transformación implican la transferencia de ADN entre bacterias individuales de la misma especie, pero ocasionalmente puede ocurrir
transferencia entre individuos de diferentes especies bacterianas, y esto puede tener consecuencias importantes, como la transferencia de resistencia a los antibióticos.
Muchas bacterias son móviles (capaces de moverse por sí mismas) y lo hacen mediante una variedad de mecanismos. Los mejor estudiados son los flagelos, largos filamentos que son girados por un motor en la base para generar un movimiento similar a una hélice. El flagelo bacteriano está formado por aproximadamente 20
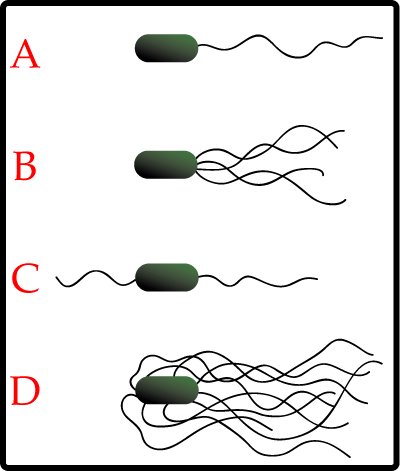
proteínas, y se requieren aproximadamente otras 30 proteínas para su regulación y ensamblaje
Las bacterias pueden utilizar los flagelos de diferentes maneras para generar diferen- tes tipos de movimiento. Muchas bacterias (como E. coli) tienen dos modos distintos de movimiento: movimiento hacia adelante (nadar) y volteretas. La caída les permite reo- rientarse y hace que su movimiento sea un paseo aleatorio tridimensional
Otros dos tipos de movimiento bacteriano se denominan motilidad de contracción , que se basa en una estructura llamada pilus tipo IV
En la motilidad espasmódica, el pilus en forma de bastón se extiende fuera de la célula, se une a algún sustrato y luego se retrae, empujando la célula hacia adelante. Las bacterias móviles son atraídas o repelidas por ciertos estímulos en comportamientos llamados impuestos: estos incluyen quimiotaxis, fototaxis, taxis energéticos y magnetotaxis

Algunas bacterias tienen sistemas químicos que generan luz. Esta bioluminiscencia ocurre a menu- do en bacterias que viven en asociación con peces, y la luz probablemente sirve para atraer peces u otros animales grandes
Las bacterias a menudo funcionan como agregados multicelulares conocidos como biopelículas, intercambiando una variedad de señales moleculares para la comunicación entre células y participando en un comporta- miento multicelular coordinado
Los beneficios comunitarios de la cooperación multicelular incluyen una división celular del trabajo, el acceso a recursos que las células individuales no pueden utilizar de manera efectiva, la defensa colectiva contra los antagonistas y la optimización de la supervivencia de la población al diferenciarse en distintos tipos de células. Por ejemplo, las bacterias en biopelículas pueden tener una resistencia más de quinientas veces mayor a los agentes antibacterianos que las bacterias "planctónicas" individuales de la misma especie.
Un tipo de comunicación intercelular mediante una señal molecular se llama quorum sensing, que sirve para determinar si la densidad de población local es suficiente para respaldar la inversión en procesos que sólo tienen éxito si un gran número de organismos similares se comportan de manera similar, como la excreción de enzimas digestivas.
En la clasificación científica establecida por Carl Linnaeus, cada especie se asigna a un género dando como resultado un nombre de dos partes. Este nombre denota los dos niveles más bajos en una jerarquía de rangos, agrupaciones de especies cada vez más grandes basadas en rasgos comunes. De estos rangos, los dominios son el nivel de categorización más general. Actualmente, los científicos clasifican toda la vida en sólo tres dominios: eucariotas, bacterias y arqueas
La taxonomía bacteriana es la clasificación de cepas dentro del dominio Bacteria en jerarquías de similitud. Esta clasificación es similar a la de plantas , mamíferos y otras taxonomías. Sin embargo, los biólogos especializados en diferentes áreas han desarrollado diferentes convenciones taxonómicas a lo largo del tiempo.
La clasificación busca describir la diversidad de especies bacterianas nombrando y agrupando organismos basándose en similitudes. Las bacterias se pueden clasificar según la estructura celular, el metabolismo celular o según las diferencias en los componentes celulares, como el ADN, los ácidos grasos, los pigmentos, los antígenos y las quinonas.
La clasificación bacteriana moderna enfatiza la sistemática molecular, utilizando técnicas genéticas como la determinación de la relación guanina citosina, la hibridación genoma-genoma, así como la secuenciación de genes que no han sufrido una transferencia genética lateral extensa, como el gen rRNA
La tinción de Gram, desarrollada en 1884 por Hans Christian Gram, caracteriza las bacterias en función de las características estructurales de sus paredes celulares. Las capas gruesas de peptidoglicano en la pared celular "Grampositiva" se tiñen de color púrpura, mientras que la delgada pared celular "Gramnegativa" aparece rosada. Al combinar la morfología y la tinción de Gram, la mayoría de las bacterias se pueden clasificar como pertenecientes a uno de cuatro grupos (cocos Gram positivos, bacilos Gram positivos, cocos Gram negativos y bacilos Gram negativos). Algunos organismos se identifican mejor mediante otras tinciones, en particular micobacterias o Nocardia. Es posible que sea necesario identificar otros organismos mediante su crecimiento en medios especiales o mediante otras técnicas, como la serología
Los diagnósticos que utilizan herramientas basadas en el ADN, como la reacción en cadena de la polimerasa, son cada vez más populares debido a su especificidad y velocidad, en comparación con los métodos basados en cultivos. Sin embargo, el número total de especies bacterianas no se conoce y ni siquiera se puede estimar con certeza. Según la clasificación actual, hay poco menos de 9.300 especies conocidas de procariotas, que incluyen bacterias y arqueas.
En el siguiente objeto interactivo, haz clic sobre la imagen para observar algunas especies de bacterias.
Iniciamos este apartado con el siguiente reporte de One Health Trust:
Fue un verdadero misterio. Las personas que acudieron a hospitales y clínicas de todo Estados Unidos estaban infectadas con una cepa rara y preocupante de bacteria que era resistente a la mayoría de los medicamentos utilizados para tratarla.
La cepa en sí nunca se había visto antes. Se trataba de Pseudomonas aeruginosa, nada raro en sí mismo. Pero este tenía un cambio genético que le permitió librarse de los efectos incluso de los antibióticos más fuertes que se suelen utilizar para tratarlo. ¿De dónde había venido?
Fue necesario mucho trabajo de detective, pero los Centros para el Control y la Prevención de Enfermedades de EE. UU. y los funcionarios de salud estatales finalmente descubrieron que la fuente común eran las gotas para los ojos. Localizaron un par de marcas de lubricantes para ojos y contuvieron el brote.
Ahora están trabajando para descubrir cómo llegó este germen en particular a las gotas para los ojos y cómo adquirió las mutaciones que lo hicieron tan resistente al tratamiento. Y están trabajando para asegurarse de que no siga acechando en los cuerpos de personas desprevenidas.
El cuerpo está continuamente expuesto a muchas especies de bacterias, incluidas las comensales beneficiosas, que crecen en la piel y las membranas mucosas, y las saprófitas, que crecen principalmente en el suelo y en la materia en descomposición. La sangre y los fluidos tisulares contienen nutrientes suficientes para
sostener el crecimiento de muchas bacterias. El cuerpo tiene mecanismos de defensa que le permiten resistir la invasión microbiana de sus tejidos y le otorgan una inmunidad natural o resistencia innata contra muchos microorganismos.
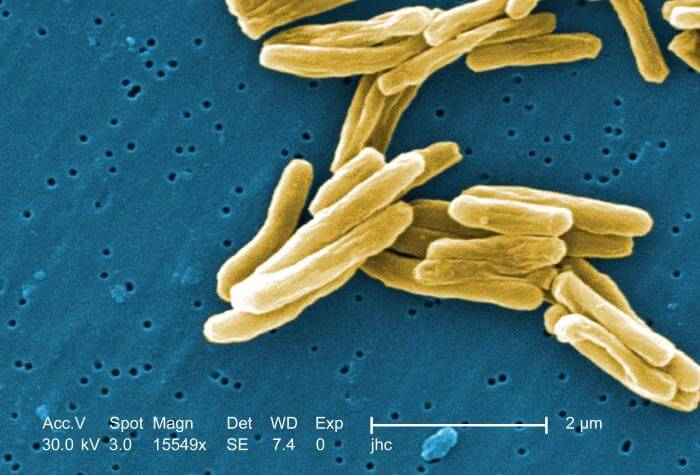
Las bacterias patógenas son una de las principales causas de muerte y enfermedades humanas y causan infecciones como el tétanos (causada por Clostridium tetani), fiebre tifoidea, difteria, sífilis, cólera, enfermedades transmitidas por alimentos, lepra (causada por Mycobacterium leprae) y tuberculosis (causada por por Mycobacterium tuberculosis). La causa patogénica de una enfermedad médica conocida sólo puede descubrirse muchos años después, como fue el caso del Helicobacter pylori y la úlcera péptica
Cada especie de patógeno tiene un espectro característico de interacciones con sus huéspedes humanos. Algunos organismos, como los estafilococos o los estreptococos, pueden provocar infecciones cutáneas, neumonía, meningitis y sepsis, una respuesta inflamatoria sistémica que produce shock, vasodilatación masiva y muerte
Rickettsias, que son parásitos intracelulares obligados capaces de crecer y reproducirse sólo dentro de las células de otros organismos. Una especie de Rickettsia causa tifus, mientras que otra causa la fiebre maculosa de las Montañas Rocosas. La Chlamydia o Clamidia, otro filo de parásitos intracelulares obligados, contiene especies que pueden causar neumonía o infección del tracto urinario y pueden estar involucradas en enfermedades coronarias
Las infecciones bacterianas pueden tratarse con antibióticos, que se clasifican como bactericidas si matan las bacterias o bacteriostáticos si sólo impiden el crecimiento bacteriano. Hay muchos tipos de antibióticos y cada clase inhibe un proceso que es diferente en el patógeno que el que se encuentra en el huésped. Un ejemplo de cómo los antibióticos producen toxicidad selectiva son el cloranfenicol y la puromicina, que inhiben el ribosoma bacteriano, pero no el ribosoma eucariótico estructuralmente diferente
medidas antisépticas, como esterilizar la piel antes de perforarla con la aguja de una jeringa, y mediante el cuidado adecuado de los catéteres permanentes. Los instrumentos quirúrgicos y dentales también se esterilizan para evitar la contaminación por bacterias. Los desinfectantes como la lejía se utilizan para matar bacterias u otros patógenos en las superficies para prevenir la contaminación y reducir aún más el riesgo de infección
En el siguiente objeto interactivo, puedes observar algunas bacterias patógenas (imágenes del INSTT):
En 2017, la Organización Mundial de la Salud (OMS) publicó una lista de patógenos prioritarios resistentes a los antibióticos, incluyendo las bacterias más peligrosas para la salud humana.
En la lista se pone de relieve especialmente la amenaza que suponen las bacterias gramnegativas resistentes a múltiples antibióticos. Estas bacterias tienen la capacidad innata de encontrar nuevas formas de resistir a los tratamientos y pueden transmitir material genético que permite a otras bacterias hacerse farmacorresistentes.
Esta es una parte de la lista publicada:
Responde a las preguntas del juego ¿Quién quiere ser millonario?
Diagrama de Escherichia coli (Centro para el Control y la Prevención de Enfermedades de Estados Unidos, en Wikipedia).
Bacterias escherichia coli generadas por la IA DALL-E 3.

Escherichia coli, comúnmente abreviada como E. coli, es una bacteria que se encuentra de forma natural en el intestino del ser humano y de los animales de sangre caliente. La mayoría de las cepas de E. coli son inofensivas, sin embargo, algunas, como la E. coli productora de toxina ShigaLas toxinas Shiga son una familia de toxinas relacionadas con dos grupos principales, Stx1 y Stx2, cuyos genes se considera que son parte del genoma de los profagos lambdoide. Las toxinas son llamadas Shiga por Kiyoshi Shiga, que fue el primero en describir el origen bacteriano de la disentería causada por Shigella dysenteriae. El origen más común de toxinas Shiga son las bacterias S. dysenteriae y el grupo Shigatoxigénico de Escherichia coli (STEC), el cual incluye el serotipo O157:H7 y otras E. coli enterohemorrágicas
Algunas personas, como los adultos mayores, los niños menores de 5 años, las personas con el sistema inmunitario debilitado y aquellas que viajan a ciertos países, tienen un mayor riesgo de contraer una infección por E. coli. Los síntomas de la infección por E. coli pueden incluir diarrea, cólicos estomacales y, en casos graves, insuficiencia renal. La mayoría de los casos de infección por E. coli mejoran sin tratamiento en un plazo de cinco a 10 días. Para prevenir la infección,
se recomienda aplicar medidas de control en todas las etapas de la cadena alimentaria, así como practicar una buena higiene de los alimentos y evitar el consumo de agua no tratada
Hacemos copia de esta artículo, publicado por Brenda Terrazas en Global REVISTA

En 1884, el microbiólogo y pediatra alemán Theodor Escherich comenzó un estudio de microbios intestinales infan- tiles y su papel en la digestión y la enfermedad. Durante este estudio, descubrió una bacteria de crecimiento rápido al que llamó Bacterium coli commune, pero que ahora se conoce como la estrella de rock biológica Escherichia coli.
E. coli, una bacteria en forma de bacilo (con forma de barra) gramnegativa, mide solo 1 µm de longitud por 0.35 µm de ancho (aunque esto varía dependiendo de la cepa). Es aerobia facultativa (es decir, puede sobrevivir en presencia o no de oxígeno), esto es porque puede activar o reprimir las enzimas metabólicas requeridas, dependiendo de los niveles de oxígeno. Los componentes
básicos de E. coli consisten en aproximadamente 55% de proteínas, 25% de ácidos nucleicos, 9% de lípidos, 6% de pared celular, 2.5% de glucógeno y 3% de otros metabolitos.
Presenta, además del cromosoma, plásmidos (se encuentran esencialmente en todas las bacterias) y son la base de la ingeniería genética, la clonación y secuenciación, la generación de organismos mutantes y otras muchas aplicaciones en biología molecular. Los plásmidos de manera natural, juegan un papel importante en la adaptación y evolución de estos organismos.
Su ascenso meteórico en la biología, se debe a lo fácil que resulta encontrarlo y trabajar con ella. Es principalmente parte de la microbiota intestinal de mamíferos, pero también se encuentra con menos frecuencia en micro- biomas intestinales de aves, reptiles y peces, así como en el suelo, agua, plantas y alimentos. Sin embargo, se han reportado muchas enferme- dades oportunistas peligrosas e incluso mortales cuando E. coli se establece fuera del intestino. Además de que sus cepas (organismo que presenta un fenotipo característico reproducible de una generación a la otra) resistentes, no patógenas y versátiles crecen rápidamente en muchos nutrientes diferentes (por lo que su producción puede ser a escala industrial) y es posible aislarla de prácticamente cualquier humano.
Su población varía por su constante interacción con el microbioma y con el vasto viroma (la suma total de virus existentes dentro o sobre un organismo) del huésped, contra el cual se defiende con enzimas de restricción, dichas enzimas son herramientas súper útiles en biología molecular. En consecuencia, cuando los microbiólogos de principios del siglo XX buscaban un organismo modelo, E. coli era una de las opciones más ampliamente disponibles y ahora es la piedra angular de muchos hallazgos importantes en biotecnología y muchas otras áreas. Por ejemplo:
el operón’, que es una unidad de transcripción regulada coordinadamente en bacterias. Modelo propuesto por Jacob, Monod y Wollman basado en sus estudios genéticos y bioquímicos sobre las mutaciones de E. coli;Actualmente E. coli es de los organismos más estudiados, se sabe mucho sobre su metabolismo, regulación y fisiología, y varias cepas se consideran de nivel 1 de bioseguridad.
El resultado, el avance sin precedentes en los campos de ingeniería genética académica y comercial, producción farmacéutica, evolución microbiana experimental, y desarrollos biotecnológicos. No es exagerado decir que E. coli es ahora el organismo modelo más importante en muchas áreas de investigación.

Theodor Escherich (29 de noviembre de 1857 – 15 de febrero de 1911) fue un destacado pediatra, bacteriólogo y profesor austroalemán. Nació en Ansbach, Reino de Baviera, y falleció en Viena, Austria-Hungría. Es conocido por su descubrimiento en 1885 de la bacteria Escherichia coli, la cual fue bautizada en su honor póstumamente en 1919.
Escherich recibió su doctorado en medicina en 1881 y se desempeñó como profesor en varias universidades, incluyendo la Universidad de Múnich, Graz y Viena. En 1902, se convirtió en profesor de pediatría en Viena, donde dirigió el Hospital de Niños de Santa Ana. Fue un pionero en el uso de los rayos X como herramienta de diagnóstico en niños y un defensor de la importancia de la prevención en la medicina. Su energía para el trabajo era tremenda, y su disposición para el trabajo, vigorosa y magistral. Escherich fue descrito como impulsivo, estricto consigo mismo pero amable con los demás. Su reputación profesional era internacional, y su carrera es un admirable ejemplo de la vida de este médico, científico y profesor universitario (perplexity.ai).
Theodor Escherich es considerado el fundador de la pediatría moderna.
“… Theodor Escherich publicó los resultados de sus investigaciones en numerosas revistas médicas, fue miembro de muchas Sociedades Científicas y se le conocía como consultor pediátrico en toda Europa. Su reputación profesional era internacional. Su energía era tremenda y su disposición para el trabajo vigorosa y magistral. Se le ha descrito como impulsivo, con una precisión fuera de lo común, determinado, leal, severo consigo mismo pero amable con los demás. Que los niños, sus pacientes, le querían es evidente, como lo es que él les correspondía y, por tanto, esto acredita su buen corazón. Su carrera es un admirable ejemplo de la vida de este médico, científico y profesor universitario…”
Algunas estirpes de E. coli han adquirido nuevos genes, suministrados por elementos móviles como plásmidos y virus atemperados (que se integran en el genoma de la célula) y pueden convertirlas en patógenas. Estos genes pueden codificar para moléculas de superficie que les permiten colonizar el intestino delgado, para toxinas semejantes a la del cólera o la de la disentería y para resistir a la acción de los antibióticos. Afortunadamente, estos genes nuevos suponen que la célula debe replicar más ADN que las que carecen de ellos, por lo que las estirpes patógenas crecen y se multiplican más despacio que las mutualistas y, por tanto, tienden a ser sustituidas por éstas cuando no hay condiciones que favorecen el desarrollo del patógeno (Ibid.).
Theodor Escherich fue sin duda el primer infectólogo pediátrico

Una bacteria de importancia clínica es aquella que puede causar una enfermedad o una infección en el ser humano, y que requiere de un diagnóstico y un tratamiento adecuados. Algunos ejemplos de bacterias de importancia clínica son: Streptococcus, Staphylococcus, Yersinia y Escherichia coli. Estas bacterias pueden provocar diversas patologías, como faringitis, neumonía, septicemia, peste, diarrea, entre otras.
Para identificar una bacteria de importancia clínica, se utilizan diferentes métodos, como la observación al microscopio, el cultivo, la tinción, las pruebas bioquímicas y las técnicas de biología molecular. El conocimiento de la estructura, el metabolismo, los factores de virulencia y la sensibilidad a los antibióticos de las bacterias de importancia clínica es fundamental para comprender la fisiopatología, la epidemiología y el tratamiento de las enfermedades infecciosas.
Por otra parte, los microorganismos más frecuentemente implicados en los brotes epidémicos son: Staphylococcus aureus resistente a meticilina, Enterococcus spp. resistente a glucopéptidos, Klebsiella pneumoniae productora de BLEEs, Acinetobacter baumannii y Pseudomonas aeruginosa multirresistentes y, en la última década, Enterobacterias productoras de carbapenemasas y/o resistentes a carbapenémicos. Existen cepas de E. coli que son resistentes a uno o más antibióticos, que obliga a identificar el origen de cualquier brote que se presente.
Algunos casos reportados de brotes de E. coli, son los siguientes:
Fue un brote epidémico causado por la toxi-infección de un serotipo de la bacteria Escherichia coli, el O104:H4, perteneciente a los filos Escherichia coli enterohemorrágica (EHEC) que produjo la muerte de al menos 53 personas en Alemania y más de un millar de infectados
Al parecer hubo evidencia epidemiológica de que los brotes de soja y de otras semillas (lentejas, judías azuki, alfalfa...) son el vehículo del brote en Alemania por la inusual bacteria E. coli O104:H4. Se ha apuntado como probable que la contaminación ocurrió por la ingestión de alimentos servidos en el restaurante Kartoffel-Keller de la ciudad de Lubeca, en el norte del país, en el Festival del Puerto de Hamburgo, al que asistieron casi un millón de personas, y últimamente en otros 20 restaurantes de la zona que tenían como denominador común que los pacientes afectados habían consumido en los mismos brotes de semillas. Se ha establecido que las personas que comieron en esos restaurantes tenían 9 veces más probabilidades de contraer la enfermedad, lo cual es una evidencia epidemiológica de peso (Wikipedia).

Ilustración creada por la IA Firefly de Adobe
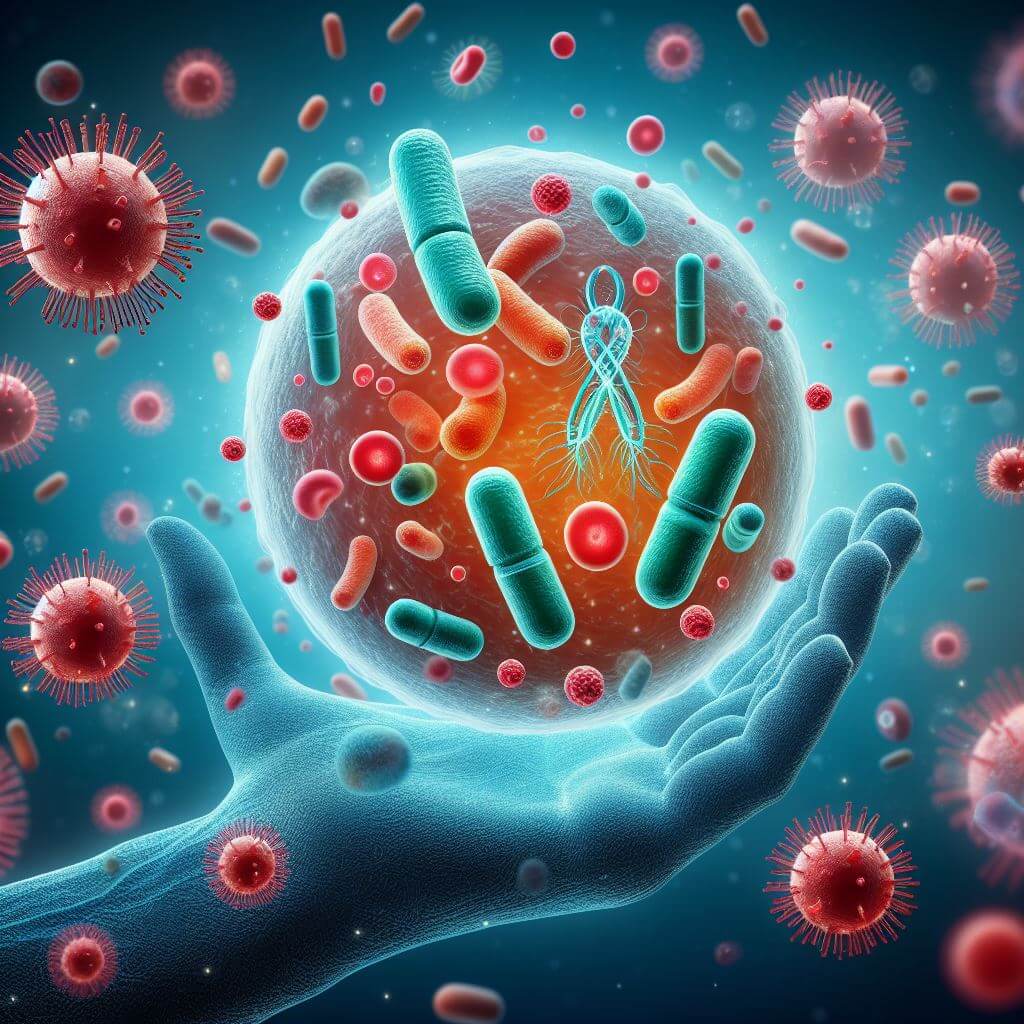
Solo un organismo, Staphy- lococcus aureus, se asoció con más de 1 millón de muertes en 2019 (1 105 000 muertes). Cuatro patógenos adicionales se asociaron con más de 500 000 muertes cada uno en 2019; éstas fueron Escherichia coli, S pneumoniae, Klebsiella pneumoniae y Pseudomonas aeruginosa. Estos cinco patógenos principales se asociaron con el 30,9% de todas las muertes relacionadas con infecciones y fueron responsables del 54,9% de todas las muertes entre las bacterias investigadas.
Se estimó que S aureus tenía el mayor número de muertes tanto en hombres (601 000 muertes) como en mujeres (504 000 muertes), y fue seguido de cerca por E coli, donde la diferencia en el número de muertes entre mujeres (450 000 muertes) y hombres (500 000 muertes) fue menor.
Esta investigación da cuenta de la importancia de mayores estudios sobre las infecciones bacterianas
Resistencia bacteriana. Un estudio de la Organización Mundial de la Salud (OMS), publicado el 22 de diciembre de 2022, reveló que más del 50% de las infecciones bacterianas que ponen en riesgo la vida se están volviendo resistentes a los tratamientos con antibióticos existentes. Según datos de 87 países en 2020, las bacterias que a menudo causan infecciones del torrente sanguíneo en los hospitales presentan altos niveles de resistencia al tratamiento con medicamentos antibacterianos.
“La resistencia a los antimi- crobianos socava la medicina moderna y pone en riesgo millones de vidas”, apuntó el director general de la OMS, Tedros Adhanom Ghebreyesus.
Entre otros hallazgos, el estudio subrayó que más del 60% de las infecciones por Neisseria gonorrea, una enfermedad muy común de transmisión sexual, son cada vez más resistentes a los antibióticos orales más utilizados para tratarlas. Asimismo, más del 20% de los casos de E. coli, el patógeno más común en las infecciones del tracto urinario, son resistentes a la ampicilina y al cotrimoxazol, así como a los tratamientos con fluoroquinolonas (OMS).
La bacteria E. coli es una de las principales causas de infecciones gastrointestinales en niños menores de cinco años en el mundo en desarrollo y se calcula que un 7% de la población infantil de estas regiones muere a causa de la diarrea provocada por estas enfermedades.
Según la Organización Mundial de la Salud, la diarrea -que es el principal síntoma de una infección gastrointestinal- mata cada año en el mundo en desarrollo a 2,2 millones de personas, 1,5 millones de éstas son niños menores de cinco años
La resistencia a los fármacos antibacterianos tiene particular importancia en América Latina
Los seis patógenos más mortales fueron Staphylococcus aureus, Escherichia coli, Klebsiella pneumoniae, Streptococcus pneumoniae, Pseudomonas aeruginosa y Acinetobacter baumannii, responsables de 45 mil muertes asociadas a RAM.
En Colombia, algunos estudios reportan la detección de E. coli patógenas intestinales en Colombia, siendo la E. coli enterotoxigénica la cepa más frecuentemente asociada a diarrea en niños menores de 5 años. Otros patógenos detectados en estos pacientes incluyen las E. coli enteroagregativa, enteropatógena, productora de toxina Shiga
Según el Instituto Nacional de Salud (INS), en 2018, el distrito de Barranquilla había superado la media nacional de resistencia a cefalosporinas de tercera generación en los servicios de UCI y No UCI tanto para E. coli como para K. pneumoniae, con valores promedios de 35,8% y 28% respectivamente.
Según cálculos aproximados de la OMS, la E. coli es responsable cada año de la muerte de 380.000 niños menores de cinco años en el mundo.
Imagen de la Escherichia coli generada por DALL-E 3.
Bacteria escherichia coli generadas por la IA DALL-E 3.
La mayor parte de la información de este capítulo, fue tomada de HandWiki.
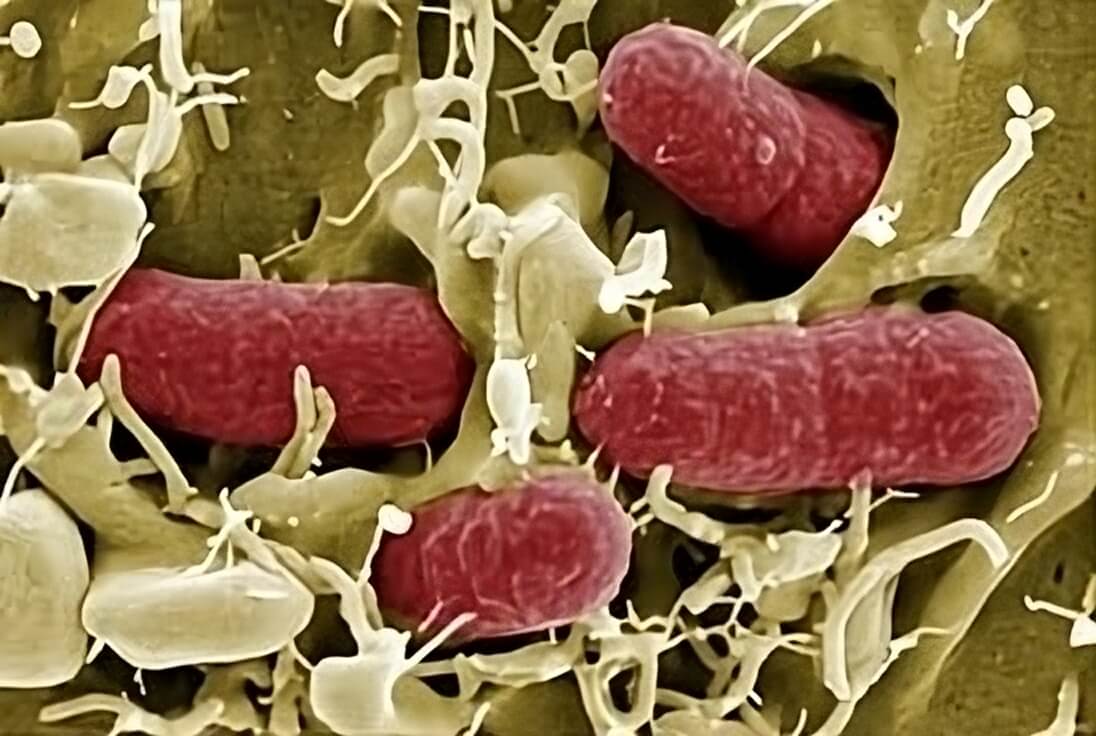
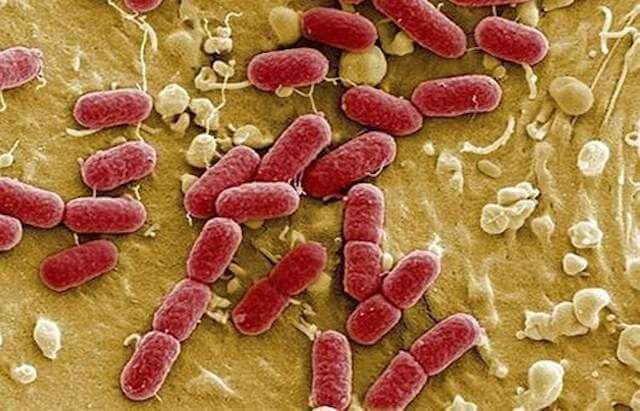
Escherichia coli es una bacteria coliforme gramnegativa, anaeróbica facultativa, en forma de bastón, que se encuentra comúnmente en el intestino inferior de los organismos de sangre caliente
E. coli y otros anaerobios facultativos constituyen aproximadamente el 0,1% de la microbiota intestinal
ambientalmente persistente que puede sobrevivir durante muchos días y crecer fuera de un huésped
La bacteria se puede cultivar de manera fácil y económica en un laboratorio, y ha sido investigada intensamente durante más de 60 años. E. coli es un quimioheterótrofo cuyo medio químicamente definido debe incluir una fuente de carbono y energía. E. coli es el organismo modelo procariótico más estudiado y una especie importante en los campos de la biotecnología y la microbiología, donde ha servido como organismo huésped para la mayoría de los trabajos con ADN recombinante. En condiciones favorables, se tarda tan sólo 20 minutos en reproducirse.
Las células suelen tener forma de bastón y miden aproximadamente 2,0 μm de largo y 0,25 a 1,0 μm de diámetro, con un volumen celular de 0,6 a 0,7 μm3. E. coli se tiñe como gramnegativa porque su pared celular está compuesta por una fina capa de peptidoglicano y una membrana externa. Durante el proceso de tinción, E. coli adquiere el color de la contratinción de safranina y se tiñe de rosa. La membrana externa que rodea la pared celular proporciona una barrera contra ciertos antibióticos, de modo que la penicilina no daña a E. coli. Los flagelos que permiten nadar a las bacterias tienen una disposición perítrica


Ilustración creada por la IA DALL-E 3
E. coli puede vivir en una amplia variedad de sustratos y utiliza fermentación ácida mixta en condiciones anaeróbicas, produciendo lactato, succinato, etanol, acetato y dióxido de carbono. Dado que muchas vías de fermentación con ácidos mixtos producen gas hidrógeno, estas vías requieren que los niveles de hidrógeno sean bajos, como es el caso cuando E. coli vive junto con organismos consumidores de hidrógeno, como los metanógenos o las bacterias reductoras de sulfato.
Además, el metabolismo de E. coli se puede reconfigurar para utilizar únicamente CO2 como fuente de carbono para la producción de biomasa. En otras palabras, el metabolismo de este heterótrofo obligado puede alterarse para mostrar capacidades autótrofas expresando heterólogamente genes de fijación de carbono, así como formiato deshidrogenasa y realizando experimentos de evolución en laboratorio. Esto se puede hacer usando formiato para reducir los portadores de electrones y suministrar el ATP requerido en las vías anabólicas dentro de estos autótrofos sintéticos
E. coli tiene tres vías glucolíticas nativas: EMPP, EDP y OPPP. El EMPP emplea diez pasos enzimáticos para producir dos piruvatos, dos ATP y dos NADH por molécula de glucosa, mientras que el OPPP
sirve como ruta de oxidación para la síntesis de NADPH. Aunque el EDP es el más termodinámicamente favorable de las tres vías, E. coli no utiliza el EDP para el metabolismo de la glucosa, basándose principalmente en el EMPP y el OPPP. El EDP permanece principalmente inactivo excepto durante el crecimiento con gluconato.
Represión de catabolitos. Cuando crecen en presencia de una mezcla de azúcares, las bacterias suelen consumir los azúcares de forma secuencial mediante un proceso conocido como represión de catabolitos. Al reprimir la expresión de los genes implicados en la metabolización de los azúcares menos preferidos, las células normalmente consumirán primero el azúcar que produce la mayor tasa de crecimiento, seguido por el azúcar que produce la siguiente tasa de crecimiento más alta, y así sucesivamente. Al hacerlo, las células se aseguran de que sus recursos metabólicos limitados se utilicen para maximizar la tasa de crecimiento. El ejemplo más utilizado de esto con E. coli implica el crecimiento de la bacteria en glucosa y lactosa , donde E. coli consumirá glucosa antes que lactosa. También se ha observado la represión de catabolitos en E. coli en presencia de otros azúcares distintos de la glucosa, como arabinosa y xilosa, sorbitol, ramnosa y ribosa. En E. coli, la represión del catabolito de la glucosa está regulada por el sistema fosfotransferasa, una cascada de fosforilación de múltiples proteínas que acopla la captación y el metabolismo de la glucosa

Ilustración creada por la IA Leonardo
Crecimiento. El crecimiento óptimo de E. coli se produce a 37 °C (99 °F), pero algunas cepas de laboratorio pueden multiplicarse a temperaturas de hasta 49 °C (120 °F)
Adaptación genética. E. coli y las bacterias relacionadas poseen la capacidad de transferir ADN mediante conjugación o transducción bacteriana, lo que permite que el material genético se propague horizontalmente a través de una población existente. El proceso de
transducción, que utiliza el virus bacteriano llamado bacteriófago
E. coli abarca una enorme población de bacterias que exhiben un grado muy alto de diversidad tanto genética como fenotípica. La secuenciación del genoma de muchos aislados de E. coli y bacterias relacionadas muestra que sería deseable una reclasificación taxonómica. Sin embargo, esto no se ha hecho, en gran parte debido a su importancia médica, por lo tanto E. coli sigue siendo una de las especies bacterianas más diversas: sólo el 20% de los genes en un genoma típico de E. coli se comparte entre todas las cepas
De hecho, desde un punto de vista más constructivo, los miembros del género Shigella (S. Dysenteriae, S. flexneri, S. boydii y S. sonnei) deberían clasificarse como cepas deE. coli, un fenómeno denominado taxones disfrazados
Una cepa es un subgrupo dentro de la especie que tiene características únicas que lo distinguen de otras cepas. Estas diferencias suelen ser detectables sólo a nivel molecular; sin embargo, pueden provocar cambios en la fisiología o el ciclo de vida de la bacteria. Por ejemplo, una cepa puede adquirir capacidad patógena, la capacidad de utilizar una fuente única de carbono, la capacidad de ocupar un nicho ecológico particular o la capacidad de resistir agentes antimicrobianos. Las diferentes cepas de E. coli suelen ser específicas del huésped, lo que permite determinar la
fuente de contaminación fecal en muestras ambientales. Por ejemplo, saber qué cepas de E. coli están presentes en una muestra de agua permite a los investigadores hacer suposiciones sobre si la contaminación se originó en un ser humano, otro mamífero o un ave.
Un sistema de subdivisión común de E. coli, pero que no se basa en la relación evolutiva, es por serotipo, que se basa en los principales antígenos de superficie (antígeno O: parte de la capa de lipopolisacárido; H: flagelina; antígeno K : cápsula), por ejemplo, O157:H7)
Como todas las formas de vida, las nuevas cepas de E. coli evolucionan a través de procesos biológicos naturales de mutación, duplicación de genes y transferencia horizontal de genes. En particular, el 18% del genoma de la cepa de laboratorio MG1655 se adquirió horizontalmente desde la divergencia con Salmonella
sanos, pero que suele ser letal para los niños del mundo en desarrollo. Cepas más virulentas, como O157:H7, causan enfermedades graves o la muerte en personas mayores, muy jóvenes o inmuno- comprometidas
Los géneros Escherichia y Salmonella divergieron hace unos 102 millones de años, un evento no relacionado con la divergencia mucho anterior de sus huéspedes: el primero se encuentra en mamíferos y el segundo en aves y reptiles
Los experimentos de evolución a largo plazo utilizando E. coli, iniciados por Richard Lenski en 1988, han permitido la observación directa de la evolución del genoma a lo largo de más de 65.000 generaciones en el laboratorio. Por ejemplo, E. coli normalmente no tiene la capacidad de crecer aeróbicamente con citrato como fuente de carbono, que se utiliza como criterio de diagnóstico para diferenciar E. coli de otras bacterias estrechamente relacionadas, como Salmonella. En este experimento, una población de E. coli desarrolló inesperadamente la capacidad de metabolizar el citrato aeróbicamente, un cambio evolutivo importante con algunas características de especiación microbiana.
En el mundo microbiano se puede establecer una relación de depredación similar a la observada en el mundo animal, se ha visto que E. coli es presa de múltiples depredadores generalistas, como Myxococcus xanthus. En esta relación depredador-presa se observa una evolución paralela de ambas especies a través de modificaciones genómicas y fenotípicas, en el caso de E. coli las modificaciones se obtienen en dos aspectos implicados en su virulencia como es la producción mucoide (producción excesiva de alginato ácido exoplásmico) y la supresión del gen OmpT, produciendo en generaciones futuras una mejor adaptación de una de las especies que se ve contrarrestada por la evolución de la otra, siguiendo un modelo coevolutivo demostrado por la hipótesis de la Reina RojaLa hipótesis de la Reina Roja (también conocida como el efecto Reina Roja, la carrera de la Reina Roja o la dinámica de la Reina Roja) es una hipótesis evolutiva de 1973 que propone que los organismos (entendidos como poblaciones o especies) deben adaptarse, evolucionar y proliferar constantemente para sobrevivir mientras compiten con otros organismos en continua evolución, en un entorno además en constante cambio, y conseguir así una ventaja reproductiva frente a sus rivales. En otras palabras, dicha hipótesis describe la necesaria adaptación continua de las especies solo para mantener el statu quo (estado del momento actual) con su entorno. El término está tomado de la novela de Lewis Carroll Alicia a través del espejo, donde los habitantes del país de la Reina Roja deben correr lo más rápido que puedan, solo para permanecer donde están, pues el país se mueve con ellos ( Wikipedia).
Las técnicas para la secuenciación del genoma completo han permitido que se encuentren disponibles genomas de E. coli de diferentes filogrupos y patotipos, permitiendo establecer un genoma núcleo (core en inglés) de E. coli. Esto permitió comparar los genomas de cepas patógenas contra el de las comensales, encontrando que en promedio, el genoma de las cepas patógenas contiene un millón más de pares de bases más, dentro de estas regiones se han localizado las secuencias asociadas a factores de virulencia, resistencia y aptitud que contribuyen a la patogenicidad de E. coli ( Wikipedia).
Las cepas patogénicas de E. coli productoras de diarrea en el hombre se han clasificado según el factor genético que define su virulencia y que contribuye al cuadro clínico asociado, definiendo a diferentes tipos patogénicos, conocidos como patotipos o virotipos; de esta forma se han definido al menos siete tipos de patotipos intestinales son: E. coli enteropatogénica (EPEC), E. coli enterotoxigénica (ETEC), E. coli enteroinvasiva (EIEC), E. coli enteroagregativa (EAEC), E. coli productora de toxina Shiga (STEC), E. coli de adherencia difusa (ADEC) y E. coli enterohemorrágica (EHEC). El patotipo EIEC se ha asociado con la enfermedad de Crohn, mientras que el patotipo DAEC se asocia con colitis ulcerativa ECEAH) que lleva genes de virulencia de la EAEC y la ECST. Las cepas de E. coli son altamente heterogéneas y continuaran adaptándose.
La cepa capaz de provocar enfermedad fuera del tracto intestinal, se denomina E. coli patógena extraintestinal (ExPEC), son responsables de una variedad de enfermedades, la más común es la infección de vías urinarias, causadas por cepas de E. coli uropatogénica (UPEC), pero pueden colonizar el tracto respiratorio, el sistema nervioso central (E. coli asociada a meningitis neonatal, NMEC) y otras infecciones ya sea generalizadas en bacteriemias, o en diversas ubicaciones (articulaciones, globos oculares, glándulas, peritoneo, hígado, hueso, cerebro, corazón, próstata, venas y otros). Estas cepas pueden ser portadas por animales, tales como las aves de crianza, cerdos, ganado vacuno, animales de compañía, de tal forma que la enfermedad extraintestinal puede ser adquirida por el contacto con estos animales o sus subproductos. Una cepa en particular, produce brotes de enfermedad relevantes en la industria de las aves y se denomina E. coli patógena aviar (APEC). Todos estos tipos, comparten
diversos factores de virulencia que les permiten adherirse a las células infectadas (p. ej., fimbrias tipo 1 y fimbrias tipo P), factores que les permiten evitar o sobrevivir a los sistemas de defensa del huésped (como cápsulas y lipopolisacáridos), mecanismos de adquisición de nutrientes (sideróforos), proteasas, invasinas y toxinas (hemolisina y factor citotóxico necrosante)
En el siguiente objeto interactivo, pon las imágenes en el contenedor correspondiente:
Escherichia coli enterotoxigénica (ETEC) es un tipo de Escherichia coli y una de las principales causas bacterianas de diarrea en el mundo en desarrollo
La infección por ETEC puede provocar diarrea acuosa profusa sin sangre ni leucocitos y calambres abdominales. También pueden producirse fiebre, náuseas con o sin vómitos, escalofríos, pérdida de apetito, dolor de cabeza, dolores musculares e hinchazón, pero son menos comunes.
La Escherichia coli enteroinvasiva (EIEC) es un tipo de bacteria patógena cuya infección provoca un síndrome idéntico a la shigelosis, con diarrea profusa y fiebre alta. Los EIEC son altamente invasivos y utilizan proteínas adhesinas para unirse a las células intestinales y entrar en ellas. No producen toxinas, pero dañan gravemente la
pared intestinal mediante la destrucción mecánica de las células.
Las EIEC están estrechamente relacionadas con Shigella, como todas las E. coli
Después de que la cepa de E. coli penetra a través de la pared epitelial, la cepa se multiplica utilizando la maquinaria de la célula huésped y se extiende a la célula epitelial adyacente. Aunque es una enfermedad invasiva, la invasión no suele traspasar la capa submucosa.
La disentería causada por EIEC generalmente ocurre entre 12 y 72 horas después de la ingestión de alimentos contaminados. La enfermedad se caracteriza por calambres abdominales, diarrea, vómitos, fiebre, escalofríos y malestar generalizado. La disentería causada por este organismo generalmente es autolimitada y no se conocen complicaciones. Actualmente se desconoce qué alimentos pueden albergar EIEC, pero cualquier alimento contaminado con heces humanas de un individuo enfermo, ya sea directamente o a través de agua contaminada, podría causar enfermedades en otros. Los brotes se han asociado con la carne de hamburguesa y la leche no pasteurizada.
Escherichia coli enteroagregativa (EAEC) es un patotipo de Escherichia coli que causa diarrea aguda y crónica tanto en el mundo desarrollado como en desarrollo
La EAEC ahora se reconoce como un patógeno entérico emergente. En particular, se informa que las EAEC son la segunda causa más común de diarrea del viajero, solo superada por la E. coli enterotoxigénica, y una causa común de diarrea entre las poblaciones pediátricas
lisogenizada por un fago que codifica la toxina Shiga. La supuesta causa del brote fueron semillas de fenogreco germinadas.
Las cepas de EAEC son genéticamente muy heterogéneas y la identificación de factores de virulencia importantes para la patogénesis ha resultado difícil
Este microorganismo constituye la especie más vieja identificada de Escherichia coli productora de diarreas y en los estudios realizados entre 1940 y 1950 se informó que algunos serotipos O:H guardaban relación con la diarrea estival de los lactantes, los brotes de diarreas en cuneros y las epidemias de diarreas en diversas comunidades. Durante la década de los años 40, se produjeron numerosos brotes epidémicos en Inglaterra, entre los cuales se registró el ocurrido en Londres y Aberdeen, en lactantes, con una mortalidad del 50 %. Estos brotes comenzaron a disminuir a partir de 1950, sin que se conociera la causa, tanto en Europa occidental como en Norteamérica. Durante
los años 1960 solo se produjeron pequeños brotes epidémicos en Inglaterra. En los países subdesarrollados, donde la lactancia materna es elevada, los brotes de diarrea se han desplazado hacia el segundo semestre de la vida (EcuRed).
Desde finales del decenio de los 60, ECEP en su mayor parte, ha desaparecido como causa fundamental de diarrea infantil en Norteamérica y Europa, aunque sigue siendo un patógeno importante productor de diarrea en Sudamérica, África y Asia.
Microorganismo descrito por primera vez durante la aparición de brotes por la ingestión de hamburguesas no bien cocinadas y elaboradas con carne de vacuno. En un estudio realizado donde se aisló la verotoxina de la Escherichia coli enterohemorrágica (EHEC), en el que se relacionó con la ingestión de comidas contaminadas. Existen diversos serotipos pero el más estudiado y que produce cuadros más graves es el O157:H7. El período de aparición es variable (EcuRed).
La primera secuencia completa de ADN de un genoma de E. coli (cepa de laboratorio K-12 derivada MG1655) se publicó en 1997. Se trata de una molécula de ADN circular de 4,6 millones de pares de bases de longitud y que contiene 4288 genes codificadores de proteínas anotados (organizados en 2584 operones), siete operones de ARN ribosómico (ARNr) y 86 genes de ARN de transferencia (ARNt). A
pesar de haber sido objeto de intensos análisis genéticos durante unos 40 años, muchos de estos genes eran desconocidos hasta ahora. Se descubrió que la densidad de codificación era muy alta, con una distancia media entre genes de sólo 118 pares de bases. Se observó que el genoma contenía un número significativo de elementos genéticos transponibles, elementos repetidos, profagos crípticos y restos de bacteriófagos
Se conocen más de trescientas secuencias genómicas comple- tas de las especies de Escherichia y Shigella. La secuencia del genoma de la cepa tipo de E. coli se agregó a esta colección antes de 2014. La comparación de estas secuencias muestra una notable cantidad de diversidad; sólo alrededor del 20% de cada genoma representa secuencias presentes en cada uno de los aislados, mientras que alrededor del 80% de cada genoma puede variar entre los aislados.
Cada genoma individual contiene entre 4.000 y 5.500 genes, pero el número total de genes diferentes entre todas las cepas de E. coli secuenciadas (el pangenoma) supera los 16.000. Se ha interpretado que esta gran variedad de genes componentes significa que dos tercios del pangenoma de E. coli se originaron en otras especies y llegaron a través del proceso de transferencia horizontal de genes
Imagen de la Escherichia coli generada por DALL-E 3.
Bacteria escherichia coli generada por la IA DALL-E 3.
La mayoría de las cepas de E. coli son inofensivas, pero las variedades patógenas causan intoxicación alimentaria grave, shock séptico, meningitis o infecciones del tracto urinario en humanos
En este capítulo profundizamos sobre la patogenecidad de la E. coli y, además, sobre su uso en la investigación, en tanto que la E. coli se utiliza como organismo modelo para estudiar la genética, la biología molecular y la fisiología bacteriana. La E. coli es fácil de cultivar en el laboratorio y su genoma ha sido completamente secuenciado, lo que la convierte en un organismo modelo ideal para la investigación
Pocos microorganismos son tan versátiles como Escherichia coli. Miembro importante de la microflora intestinal normal de humanos y otros mamíferos, E. coli también ha sido ampliamente explotada como huésped de clonación en tecnología de ADN recombinante. Pero E. coli es más que un simple caballo de batalla de laboratorio o un habitante intestinal inofensivo; también puede ser un patógeno muy versátil y frecuentemente mortal. Varias cepas diferentes de E. coli causan diversas enfermedades intestinales y extraintestinales mediante factores de virulencia que afectan una amplia gama de procesos celulares
Las cepas patógenas de E. coli se pueden clasificar según elemen- tos que pueden provocar una respuesta inmune en animales, a saber:
Por ejemplo, la cepa E. coli EDL933 pertenece al grupo O157:H7.
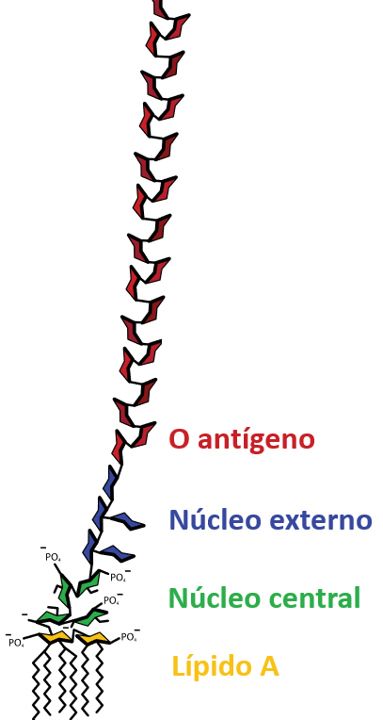
La membrana externa de una célula de E. coli contiene millones de moléculas de lipopolisacárido (LPS), que consta de:
El antígeno O se utiliza para serotipificar E. coli y estas designaciones
de grupos O van de O1 a O181, con la excepción de algunos grupos que históricamente se han eliminado, a saber, O31, O47, O67, O72, O93 (ahora K84), O94, y O122; los grupos 174 a 181 son provisionales (O174 = OX3 y O175 = OX7) o están bajo investigación (176 a 181 son STEC/VTEC). Además, existen subtipos para muchos grupos O (por ejemplo, O128ab y O128ac). Los anticuerpos contra varios antígenos O reaccionan de forma cruzada con otros antígenos O y parcialmente con antígenos K no sólo de E. coli, sino también de otras especies de Escherichia y Enterobacteriaceae
El antígeno O está codificado por el grupo de genes rfb. El gen rol (cld) codifica el regulador de la longitud de la cadena O del lipopolisacárido.
El polisacárido capsular ácido (CPS) es una capa espesa de polisacárido similar a una mucosa que rodea algunos patógenos E. coli. Hay dos grupos separados de antígenos K, denominados grupo I y grupo II (mientras que un pequeño subconjunto intermedio - K3, K10 y K54/K96 - se ha clasificado como grupo III). Los primeros (I) consisten en polisacáridos capsulares (grandes) de 100 kDa, mientras que los segundos (II), asociados con enfermedades extraintestinales, tienen un tamaño inferior a 50 kDa.
Los antígenos del grupo IK solo se encuentran con ciertos antígenos O (grupos O8, O9, O20 y O101), y se subdividen según la ausencia (IA, similar a la de las especies de Klebsiella en estructura) o la presencia (IB) de Los aminoazúcares y algunos antígenos K del grupo I están unidos al núcleo lipídico A del lipopolisacárido ( LPS K ), de manera similar a los antígenos O (y al ser estructuralmente idénticos a los antígenos O, en algunos casos solo se consideran antígenos K cuando coexpresado con otro antígeno O auténtico)
Los antígenos K del grupo II se parecen mucho a los de las bacterias grampositivas y difieren mucho en su composición y se subdividen según sus componentes ácidos; generalmente, entre el 20 y el 50% de las cadenas de CPS están unidas a fosfolípidos (Ibid.).
El antígeno H es un componente importante de los flagelos y participa en el movimiento de E. coli. Generalmente está codificado por el gen fliC. Hay 53 antígenos H identificados, numerados del H1 al H56 (H13 y H22 no eran antígenos de E. coli sino de Citrobacter freundii , y se encontró que H50 era el mismo que H10)

En humanos y animales domésticos, las cepas virulentas de E. coli causan diversas enfermedades. En humanos: gastroenteritis, infecciones del tracto urinario y meningitis neonatal. En casos más raros, las cepas virulentas también son responsables del síndrome urémico hemolítico, peritonitis, mastitis, septicemia y neumonía por gramnegativos.
Ciertas cepas de E. coli, como O157:H7, O104:H4, O121, O26, O103, O111, O145 y O104:H21, producen toxinas potencialmente letales. La intoxicación alimentaria causada por E. coli puede deberse al consumo de verduras sin lavar o de carne mal cortada y poco cocida.

Ilustración creada por la IA DALL-E 3
O157:H7 también es conocido por causar complicaciones graves e incluso potencialmente mortales, como el síndrome urémico hemolítico. Esta cepa en particular está relacionada con el brote de E. coli en Estados Unidos en 2006 debido a las espinacas frescas.
La cepa O104:H4 es igualmente virulenta. Los protocolos de tratamiento con antibióticos y de apoyo no están tan bien desarrollados (tiene la capacidad de ser muy enterohemorrágico como O157:H7, causando diarrea con sangre, pero también es más enteroagregativo, lo que significa que se adhiere bien y se acumula en las membranas intestinales). Es la cepa detrás del mortal brote de E. coli de junio de 2011 en Alemania. La gravedad de la enfermedad varía considerablemente; puede ser mortal, especialmente en niños pequeños, ancianos o personas inmunocomprometidas, pero suele ser leve. Anteriormente, los métodos higiénicos deficientes para preparar la carne en Escocia mataron a siete personas en 1996 debido al envenenamiento por E. coli y dejaron cientos más infectados.
E. coli puede albergar enterotoxinas termoestables y termolábiles. Estas últimas, denominadas LT, contienen una subunidad A y cinco subunidades B dispuestas en una holotoxina y son muy similares en estructura y función a las toxinas del cólera. Las subunidades B ayudan en la adherencia y entrada de la toxina en las células intestinales del huésped, mientras que la subunidad A se escinde e impide que las células absorban agua, provocando diarrea. La LT se secreta por la vía de secreción tipo 2
Si la bacteria E. coli escapa del tracto intestinal a través de una perforación (por ejemplo, por una úlcera, una rotura del apéndice o debido a un error quirúrgico) e ingresa al abdomen, generalmente causa una peritonitis que puede ser fatal sin un tratamiento oportuno. Sin embargo, E. coli es extremadamente sensible a antibióticos como la estreptomicina o la gentamicina. Investigaciones recientes sugieren que el tratamiento de E. coli enteropatógena con antibióticos
puede no mejorar el resultado de la enfermedad, ya que puede aumentar significativamente la posibilidad de desarrollar síndrome urémico hemolítico
La E. coli asociada a la mucosa intestinal se observa en mayor número en las enfermedades inflamatorias del intestino , la enfermedad de Crohn y la colitis ulcerosa
de la inflamación intestinal
La E. coli entérica (EC) se clasifica según sus características serológicas y propiedades de virulencia. Los principales patotipos de E. coli que causan diarrea se enumeran a continuación.
| Nombre | Hospedadores | tipo de diarrea | Descripción |
|---|---|---|---|
| E. coli entero- toxigénica (ETEC) | agente causante de la diarrea (sin fiebre) en humanos, cerdos, ovejas, cabras, ganado vacuno, perros y caballos | Acuoso | ETEC puede producir dos enterotoxinas proteicas: La mayor (la enterotoxina LT), es similar a la toxina del cólera en estructura y función. La proteína más pequeña, la enterotoxina ST, provoca la acumulación de cGMP y secreción de líquido y electrolitos en la luz intestinal. Las cepas de ETEC no son invasivas y no abandonan la luz intestinal. ETEC es la principal causa bacteriana de diarrea en niños en el mundo en desarrollo, así como la causa más común de diarrea del viajero. |
| Nombre | Hospedadores | tipo de diarrea | Descripción |
|---|---|---|---|
| E. coli entero- patógena (EPEC) | agente causante de la diarrea en humanos, conejos, perros, gatos y caballos | Acuoso | EPEC también causa diarrea, pero los mecanismos moleculares de colonización y etiología son diferentes. Los EPEC carecen de toxinas ST y LT, pero utilizan una adhesina conocida como intimina para unirse a las células intestinales del huésped. Este patotipo tiene una serie de factores de virulencia similares a los encontrados en Shigella. La adherencia a la mucosa intestinal provoca una reordenación de la actina en la célula huésped, provocando una deformación importante. Las células EPEC son moderadamente invasivas y provocan una respuesta inflamatoria. |
| E. coli entero- agregativa (EAEC) | encontrado sólo en humanos | Acuoso | Llamadas así porque tienen fimbrias que agregan células de cultivo de tejidos, las EAEC se unen a la mucosa intestinal para causar diarrea acuosa sin fiebre. EAEC no son invasivos. Producen una hemolisina y una enterotoxina ST similar a la de ETEC. |
| Nombre | Hospedadores | tipo de diarrea | Descripción |
|---|---|---|---|
| E. coli entero- invasiva (EIEC) | encontrado sólo en humanos | Acuoso | La infección por EIEC causa un síndrome idéntico a la shigelosis, con diarrea profusa y fiebre alta. |
| E. coli entero- hemorrágica (ECEH) | encontrado en humanos, ganado vacuno y cabras | Con sangre o sin sangre | El miembro más infame de este patotipo es la cepa O157:H7, que provoca diarrea con sangre y ausencia de fiebre. ECEH puede causar síndrome urémico hemolítico e insuficiencia renal repentina. Utiliza fimbrias bacterianas para su unión, es moderadamente invasiva y posee una toxina shiga codificada por fagos que puede provocar una intensa respuesta inflamatoria |
| E. coli adherente- invasiva (AIEC) | encontrado en humanos | --- | Los AIEC pueden invadir las células epiteliales intestinales y replicarse intracelularmente. Es probable que los AIEC puedan proliferar más eficazmente en huéspedes con inmunidad innata defectuosa. Se asocian con la mucosa ileal en la enfermedad de Crohn |
La transmisión de E. coli patógena a menudo ocurre mediante transmisión fecal-oral
Según la Administración de Alimentos y Medicamentos de EE. UU., el ciclo de transmisión fecal-oral puede interrumpirse cocinando adecuadamente los alimentos, previniendo la contaminación cruzada, instituyendo barreras como guantes para los trabajadores de alimentos, instituyendo políticas de atención médica para que los empleados de la industria alimentaria busquen tratamiento cuando están enfermos, pasteurización de jugos o productos lácteos y requisitos adecuados de lavado de manos.
La E. coli productora de toxina Shiga (STEC), específicamente el serotipo O157:H7, también ha sido transmitida por moscas
La E. coli uropatógena (UPEC) es responsable de aproximadamente el 90% de las infecciones del tracto urinario (ITU) observadas en
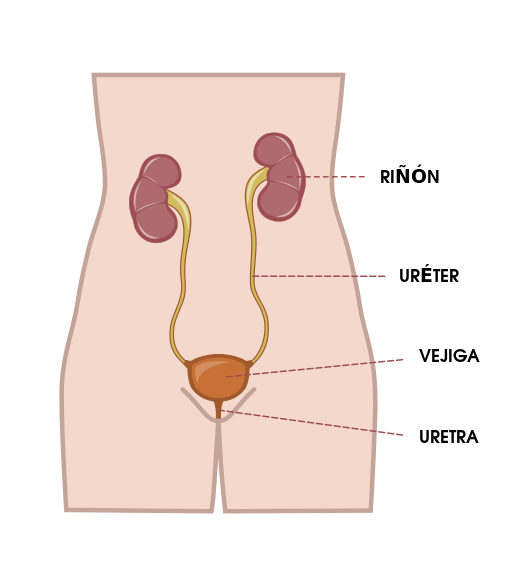
personas con anatomía normal. En las infecciones ascendentes, las bacterias fecales colonizan la uretra y se propagan por el tracto urinario hasta la vejiga y los riñones (causando pielonefritis )
La E. coli uropatógena utiliza fimbrias P (pili asociadas a pielonefritis) para unirse a las células uroteliales del tracto urinario y colonizar la vejiga. Estas adhesinas se unen específicamente a restos de D-galactosa en el antígeno del grupo sanguíneo P de los eritrocitos y las células uroepiteliales. Aproximadamente el 1% de la población humana carece de este receptor, y su presencia o ausencia dicta la susceptibilidad o no susceptibilidad de un individuo, respectivamente, a las infecciones del tracto urinario por E. coli. La E. coli uropatógena produce alfa y beta hemolisinas, que provocan la lisis de las células del tracto urinario.
Otro factor de virulencia comúnmente presente en la UPEC es la familia Dr de adhesinas, que están particularmente asociadas con cistitis y pielonefritis asociada al embarazo. Las adhesinas Dr se unen al antígeno del grupo sanguíneo Dr (Dra) que está presente en el factor acelerador de la descomposición (DAF) de los eritrocitos y otros tipos de células. Allí, las adhesinas Dr inducen el desarrollo de largas extensiones celulares que envuelven a las bacterias, acompañadas de la activación de varias cascadas de transducción de señales , incluida la activación de la quinasa PI-3.
La UPEC puede evadir las defensas inmunitarias innatas del cuerpo invadiendo células paraguas superficiales para formar comunidades bacterianas intracelulares (IBC)
Las infecciones descendentes, aunque relativamente raras, ocurren cuando las células de E. coli ingresan a los órganos del tracto urinario superior (riñones, vejiga o uréteres) desde el torrente sanguíneo.
Es producida por un serotipo de Escherichia coli que contiene un antígeno capsular llamado K1. La colonización del intestino del recién nacido con estas cepas, que están presentes en la vagina de la madre, provoca una bacteriemia que deriva en meningitis
El diagnóstico de diarrea infecciosa y la identificación de resistencia a los antimicrobianos se realiza mediante un cultivo de heces con pruebas posteriores de sensibilidad a los antibióticos. Se requieren un mínimo de 2 días y un máximo de varias semanas para cultivar patógenos gastrointestinales. Las tasas de sensibilidad (verdadero positivo) y especificidad (verdadero negativo) del cultivo de heces varían según el patógeno, aunque varios patógenos humanos no se pueden cultivar. Para muestras con cultivo positivo, la prueba de resistencia a los antimicrobianos tarda entre 12 y 24 horas adicionales en realizarse.
Las pruebas de diagnóstico molecular, en el lugar de atención, pueden identificar E. coli y la resistencia a los antimicrobianos en las cepas identificadas mucho más rápido que las pruebas de cultivo y sensibilidad. Las plataformas basadas en microarrays pueden identificar cepas patógenas específicas de E. coli y genes de RAM específicos de E. coli en dos horas o menos con alta sensibilidad y especificidad, pero el tamaño del panel de prueba es limitado.
En las muestras de heces, la microscopía mostrará bacilos gramnegativos, sin ninguna disposición celular particular. Luego, se inoculan las heces con agar MacConkey o agar EMB (o ambos). En el agar MacConkey, se producen colonias de color rojo intenso, ya que el organismo es lactosa positivo, y la fermentación de este azúcar hará que el pH del medio baje, lo que provocará un oscurecimiento del medio. El crecimiento en agar EMB produce colonias negras con un brillo metálico negro verdoso. Esto es diagnóstico de E. coli. El organismo también es lisina positivo y crece en TSI inclinado con un perfil (A/A/g+/H 2 S-). Además, IMViC es {+ + – -} para E. coli; ya que es indol positivo (anillo rojo) y rojo metilo positivo (rojo brillante), pero VP negativo (sin cambios, incoloro) y citrato negativo (sin
cambios, color verde). Las pruebas de producción de toxinas pueden utilizar células de mamíferos en cultivos de tejidos , que son rápidamente destruidas por la toxina shiga. Aunque sensible y muy específico, este método es lento y costoso
Por lo general, el diagnóstico se realiza mediante cultivo en medio sorbitol-MacConkey y luego utilizando antisuero tipificador. Sin embargo, los ensayos de látex actuales y algunos antisueros de tipificación han mostrado reacciones cruzadas con colonias distintas de E. coli O157. Además, no todas las cepas de E. coli O157 asociadas con el SUH son fermentadoras distintas del sorbitol.
El Consejo de Epidemiólogos Estatales y Territoriales recomienda que los laboratorios clínicos analicen al menos todas las heces con sangre para detectar este patógeno. Los Centros para el Control y la
Prevención de Enfermedades de EE. UU. recomiendan que "todas las heces enviadas para análisis de rutina de pacientes con diarrea aguda adquirida en la comunidad (independientemente de la edad del paciente, la estación del año o la presencia o ausencia de sangre en las heces) se cultiven simultáneamente paraE. coli O157:H7 (O157 STEC) y probado con un ensayo que detecta toxinas Shiga para detectar STEC no O157".
En el siguiente video, la Dra. Marielsa Gil, nos explica la epidemiología, patogenia, manifestaciones clínicas y tratamientos de las bacterias E. coli Enterotoxigénica y la E. coli Enterohemorrágica.
Las infecciones bacterianas generalmente se tratan con antibióticos. Sin embargo, la sensibilidad a los antibióticos de las diferentes cepas de E. coli varía ampliamente. Como organismos gramnegativos, E. coli es resistente a muchos antibióticos que son eficaces contra los organismos grampositivos. Los antibióticos que pueden usarse para tratar la infección por E. coli incluyen amoxicilina, así como otras penicilinas semisintéticas, muchas cefalosporinas, carbapenémicos, aztreonam, trimetoprim-sulfametoxazol, ciprofloxacina, nitrofuran- toína y aminoglucósidos.
La resistencia a los antibióticos es un problema creciente. Parte de esto se debe al uso excesivo de antibióticos en humanos, pero parte probablemente se deba al uso de antibióticos como promotores del crecimiento en los alimentos para animales
La E. coli resistente a los antibióticos también puede transmitir los genes responsables de la resistencia a los antibióticos a otras especies de bacterias, como Staphylococcus aureus, a través de un proceso llamado transferencia horizontal de genes. Las bacterias E. coli a menudo portan plásmidos de resistencia a múltiples medicamentos y, bajo estrés, transfieren fácilmente esos plásmidos a otras especies. La mezcla de especies en los intestinos permite que E. coli acepte y transfiera plásmidos desde y hacia otras bacterias. Por tanto, E. coli y otras enterobacterias son reservorios importantes de resistencia a los antibióticos transferibles.
La resistencia a los antibióticos betalactámicos se ha convertido en un problema particular en las últimas décadas, a medida que las cepas de bacterias que producen betalactamasas de espectro extendido se han vuelto más comunes
La creciente preocupación por la prevalencia de esta forma de "superbacteria" en el Reino Unido ha llevado a pedir una mayor vigilancia y una estrategia en todo el Reino Unido para hacer frente a las infecciones y las muertes. Las pruebas de susceptibilidad deben guiar el tratamiento en todas las infecciones en las que el organismo puede aislarse para cultivo.
La terapia con fagos (virus que atacan específicamente a bacterias patógenas) se ha desarrollado durante los últimos 80 años, principalmente en la ex Unión Soviética, donde se usaba para prevenir la diarrea


Ilustración creada por la IA DALL-E 3
causada por E. coli. Actualmente, la terapia con fagos para humanos sólo está disponible en el Centro de Terapia con Fagos en la República de Georgia y en Polonia. Sin embargo, el 2 de enero de 2007, la FDA de los Estados Unidos aprobó a Omnilytics para aplicar su fago letal E. coli O157:H7 en forma de niebla, rociado o lavado en animales vivos que serán sacrificados para el consumo humano. El fago T4 de las enterobacterias, un fago altamente estudiado, se dirige a E. coli para la infección.
Si bien la terapia con fagos como tratamiento para E. coli no está disponible en los EE. UU., algunos suplementos dietéticos disponibles comercialmente contienen cepas de fagos que se dirigen a E. coli y se ha demostrado que reducen la carga de E. coli en sujetos sanos. Sin embargo, esto no se considera terapia con fagos porque no implica la selección de fagos con actividad contra la cepa de bacteria específica de un paciente.
Los investigadores han estado trabajando activamente para desarrollar vacunas seguras y eficaces para reducir la incidencia mundial de la infección por E. coli
ya habían indicado que era seguro para los adultos. Está previsto realizar un ensayo clínico de fase III para verificar la eficacia a gran escala del tratamiento
En 2006, Fort Dodge Animal Health ( Wyeth ) introdujo una vacuna viva, atenuada y eficaz para controlar la aerosaculitis y la peritonitis en pollos. La vacuna es una vacuna avirulenta modificada genéticamente que ha demostrado protección contra O78 y cepas no tipificables.
En enero de 2007, la empresa biofarmacéutica canadiense Bioniche anunció que había desarrollado una vacuna para el ganado que reduce el número de O157:H7 eliminado en el estiércol en un factor de 1.000, a unas 1.000 bacterias patógenas por gramo de estiércol
En abril de 2009, un investigador de la Universidad Estatal de Michigan anunció que había desarrollado una vacuna funcional para una cepa de E. coli. El Dr. Mahdi Saeed, profesor de epidemiología y enfermedades infecciosas en las facultades de Medicina Veterinaria y Medicina Humana de MSU, solicitó una patente para su descubrimiento y se puso en contacto con compañías farmacéuticas para la producción comercial.]
En mayo de 2018, un equipo dirigido por investigadores de la Facultad de Medicina de la Universidad de Washington colaboró con la Universidad Johns Hopkins para realizar un estudio que profundiza en el vínculo conocido entre el tipo de sangre y la gravedad de la infección por E. coli. Los resultados del estudio mostraron que "la bacteria tiene más probabilidades de causar diarrea grave en personas con sangre tipo A", y este hallazgo puede ayudar a los esfuerzos actuales y futuros para desarrollar una vacuna eficaz contra las cepas patógenas de E. coli
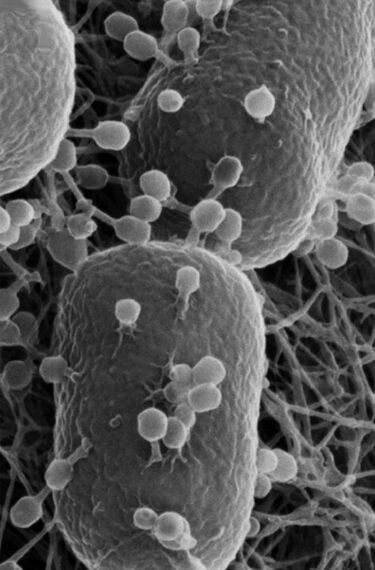
Debido a su larga historia de cultivo en laboratorio y su facilidad de manipulación, E. coli desempeña un papel importante en la ingeniería biológica y la microbiología industrial moder- nas. El trabajo de Stanley Norman Cohen y Herbert Boyer en E. coli, utilizando plásmidos y enzimas de restricción para crear ADN recombinante, se convirtió en la base de la biotecnología
E. coli es un huésped muy versátil para la producción de proteínas heterólogas
Muchas proteínas que antes se consideraban difíciles o imposibles de
expresar en E. coli en forma plegada se han expresado con éxito en E. coli. Las células de E. coli modificadas se han utilizado en el desarrollo de vacunas, biorremediación, producción de biocombustibles, iluminación y producción de enzimas inmovilizadas.
La cepa K-12 es una forma mutante de E. coli que sobreexpresa la enzima fosfatasa alcalina (ALP). La mutación surge debido a un defecto en el gen que codifica constantemente la enzima. Se dice que un gen que produce un producto sin ninguna inhibición tiene actividad constitutiva. Esta forma mutante particular se utiliza para aislar y purificar la enzima antes mencionada.
La cepa OP50 de Escherichia coli se utiliza para el mantenimiento de cultivos de Caenorhabditis elegans. La cepa JM109 es una forma mutante de E. coli que tiene deficiencia de recA y endA, se puede utilizar para la detección azul/blanca cuando las células portan el episoma del factor de fertilidad. La falta de recA disminuye la posibilidad de restricción no deseada del ADN de interés y la falta de endA inhibe la descomposición del ADN plasmídico. Por tanto, JM109 es útil para sistemas de clonación y expresión.
E. coli se utiliza frecuentemente como organismo modelo en estudios de microbiología. Las cepas cultivadas (por ejemplo, E. coli K12) están bien adaptadas al entorno del laboratorio y, a diferencia de las cepas silvestres, han perdido su capacidad de prosperar en el intestino. Muchas cepas de laboratorio pierden su capacidad de formar biopelículas
fotocatálisis. Mediante métodos estándar de recuento en placa, después de diluciones secuenciales y crecimiento en placas de gel de agar, se puede evaluar la concentración de organismos viables o UFC (Unidades formadoras de colonias) en un volumen conocido de agua tratada, lo que permite una evaluación comparativa del rendimiento de los materiales.
En 1946, Joshua Lederberg y Edward Tatum describieron por primera vez el fenómeno conocido como conjugación bacteriana utilizando E. coli como bacteria modelo, y sigue siendo el modelo principal para estudiar la conjugación. E. coli fue una parte integral de los primeros experimentos para comprender la genética de los fagos, y los primeros investigadores, como Seymour Benzer, utilizaron E. coli y el fago T4 para comprender la topografía de la estructura genética. Antes de la investigación de Benzer, no se sabía si el gen era una estructura lineal o si tenía un patrón de ramificación.
De 2002 a 2010, un equipo de la Academia de Ciencias de Hungría creó una cepa de Escherichia coli llamada MDS42, que ahora vende Scarab Genomics de Madison, WI con el nombre de "Clean Genome E. coli", donde se eliminó el % del genoma de la cepa parental (E. coli K-12 MG1655) para ayudar en la eficiencia de la biología molecular, eliminando elementos IS, pseudogenes y fagos, lo que resultó en un mejor mantenimiento de los genes tóxicos codificados por plásmidos, que a menudo son inactivados por transposones. La bioquímica y la maquinaria de replicación no se alteraron.

E. coli fue uno de los primeros organismos cuyo genoma fue secuenciado. El genoma completo de E. coli K12 fue publicado por Science en 1997
Al evaluar la posible combinación de nanotecnologías con la ecología del paisaje, se pueden generar paisajes de hábitat complejos con detalles a nanoescala
En otros estudios, la E. coli no patógena se ha utilizado como microorganismo modelo para comprender los efectos de la microgravedad simulada (en la Tierra) sobre la misma
Desde 1961, los científicos propusieron la idea de circuitos genéticos utilizados para tareas computacionales. La colaboración entre biólogos e informáticos ha permitido diseñar puertas lógicas digitales sobre el metabolismo de E. coli. Como el operón Lac que es un proceso de dos etapas, la regulación genética en las bacterias se utiliza para realizar funciones informáticas. El proceso se controla en la etapa de transcripción del ADN en ARN mensajero.
Se ha desarrollado una computadora para controlar la producción de proteínas de E.

Ilustración DALL-E 3
coli dentro de las células de levadura. También se ha desarrollado un método para utilizar bacterias para que se comporten como una pantalla LCD
En julio de 2017, experimentos separados con E. coli publicados en Nature mostraron el potencial de utilizar células vivas para tareas informáticas y almacenar información. Un equipo formado con colaboradores del Instituto de Biodiseño de la Universidad Estatal de Arizona y el Instituto Wyss de Ingeniería Biológicamente Inspirada de Harvard desarrolló una computadora biológica dentro de E. coli que respondía a una docena de entradas. El equipo llamó a la computadora "ribocomputadora", ya que estaba compuesta de ácido ribonucleico
E. coli tiene varios usos prácticos además de su uso como vector para experimentos y procesos genéticos. Por ejemplo, E. coli se puede utilizar para generar propano sintético y hormona de crecimiento humano recombinante